南湖新闻网讯(通讯员 王小斌)近日,作物遗传改良全国重点实验室、湖北洪山实验室杨宁教授、刘磊教授课题组联合深圳华大研究院刘欢研究员课题组研究成果以“A spatial transcriptome map of the developing maize ear”为题在Nature Plants发表。研究构建了首个玉米雌穗空间转录组图谱,最终共获得了12种细胞类型的转录组及其空间位置信息,鉴定并验证了4种全新的细胞类型。利用空间组学的优势,鉴定到了两个在小花分生组织顶端中特异性表达,并决定其分化确定性的MADS-box基因。通过整合单细胞转录组及空间转录组数据,研究人员探索了一种高通量基因挖掘的新方法,通过构建细胞特异性的基因共表达网络鉴定了一批潜在控制玉米雌穗建成的新基因。
玉米作为我国主要粮食和饲料来源,其产量对农业和畜牧业至关重要。提高玉米单位面积产量,特别是单穗粒数,是保障我国玉米供需平衡的关键。关键基因的时空特异性表达决定了玉米雌穗的发育,进而影响了玉米单穗产量。然而,常规的植物转录组技术往往忽略了细胞的异质性,无法获取关键基因的时空表达信息。另外,我们对于哪些细胞参类型与了玉米雌穗的形态建成仍然知之甚少。因此,当前以正向遗传学手段挖掘调控雌穗发育的相关基因,并将其应用于玉米穗部性状改良中的效率相对较低。
研究人员首先优化了Stereo-seq的实验条件,开发了适用于玉米雌穗的空间转录组测序方法,并选取了处于关键生长发育期下的6mm雌穗作为实验对象,对其分别进行空间转录组与单细胞转录组测序。在分析了空间转录组数据后,研究人员得到了12种细胞类型的转录组以及空间位置。参考已有研究中对玉米雌穗解剖学结构的描述,可以快速定义细胞群身份。最终,研究人员发现大部分细胞类型都位于已知的雌穗发育器官内,但同时也鉴定到了四种全新的细胞类型,包括三种分布在花序分生组织中的细胞和一种分布在小花分生组织中心的细胞。为了进一步验证空间转录图谱的可靠性,研究人员随机选取了在空间转录组数据中鉴定到的和已发表的具有空间表达特异性特征的标记基因(55个),利用原位杂交实验验证其表达模式。实验结果表明,74%的基因在原位杂交中呈现的表达部位与空间转录组结果一致,说明了空间转录组测序的可行性和可靠性 (图1)。
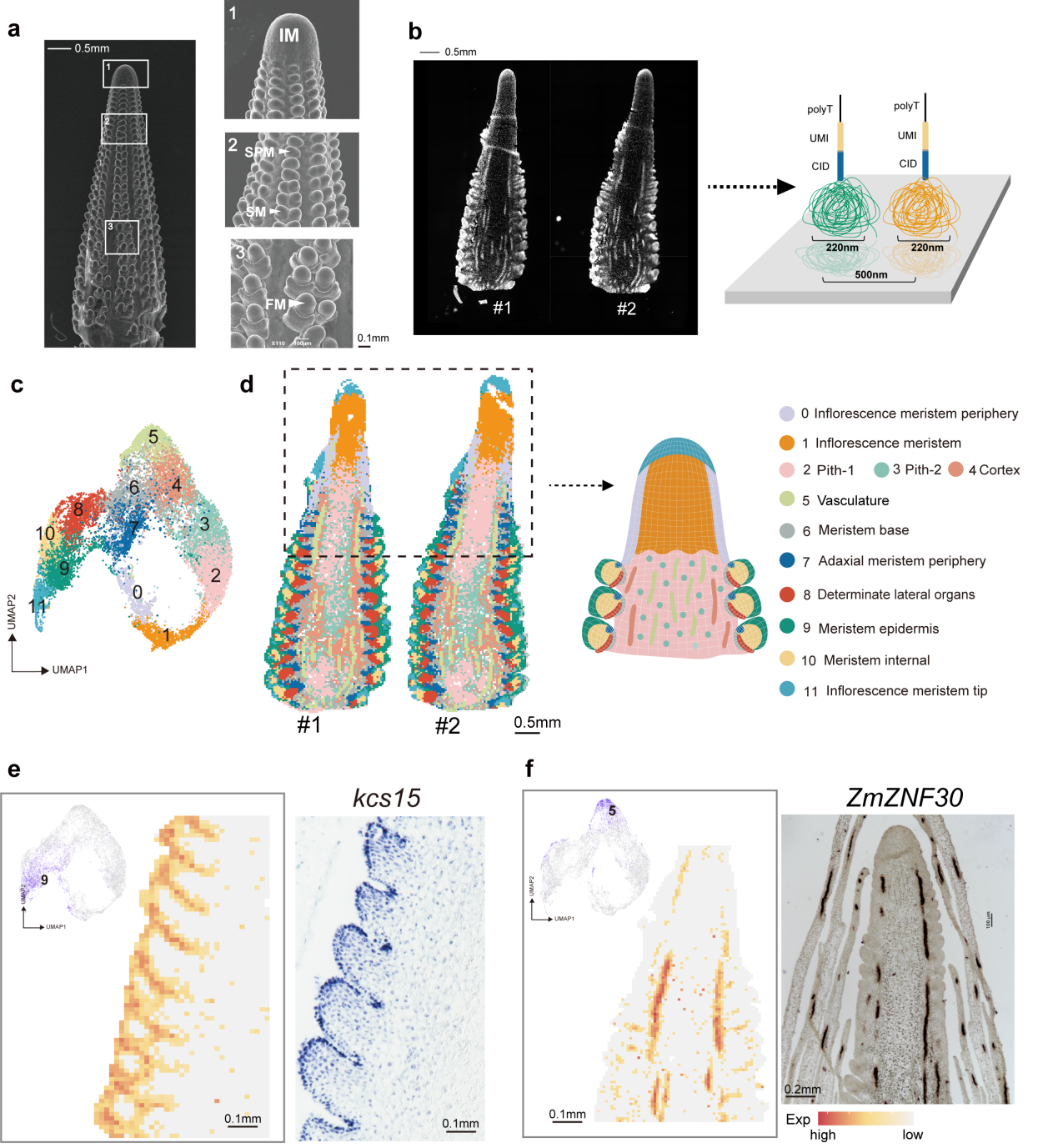
图1 构建玉米6mm雌穗的空间转录组图谱
基于Stereo-seq技术构建的6mm雌穗的空间转录组图谱能够清楚区分不同类型的分生组织。因此,根据它们各自不同的空间位置,研究人员提取了6mm雌穗的花序分生组织以及小花分生组织转录组,并进行重聚类分析。结果表明,这些分生组织可以被进一步区分为三种不同的细胞类型,分别分布在花序分生组织顶端、小花分生组织外围和小花分生组织顶端。通过检测了在三种细胞类型之间存在的一系列差异表达基因,其中包括两个MADS-box基因,ZmMADS8及其同源基因ZmMADS14,这两个MADS-box基因在DMT中特异性表达,其表达模式通过mRNA原位杂交得到了验证。先前的研究表明,MADS-box家族基因在植物器官发育中发挥了重要作用。因此,为验证ZmMADS8与ZmMADS14的功能,研究人员通过CRISPR/Cas9基因编辑技术产生了ZmMADS8和ZmMADS14的双基因敲除突变体。观察发现,在ZmMADS8和ZmMADS14的单突变体中,小花分生组织均可发育正常,能够产生正常的上位小花。而在CR-Zmmads8/14双突变体中,小花分生组织不会向下一级花器官分化,而是会逆转为不确定性长分枝,无法产生正常的花器官(图2)。这说明ZmMADS8和ZmMADS14是小花分生组织命运决定的关键因子。这些结果表明,基于Stereo-seq技术获得的玉米雌穗空间转录组数据能够区分高度相似的分生组织类型,使我们能够进一步研究分生组织在分化过程中产生的细微变化,并有助于鉴定关键的发育调控因子。
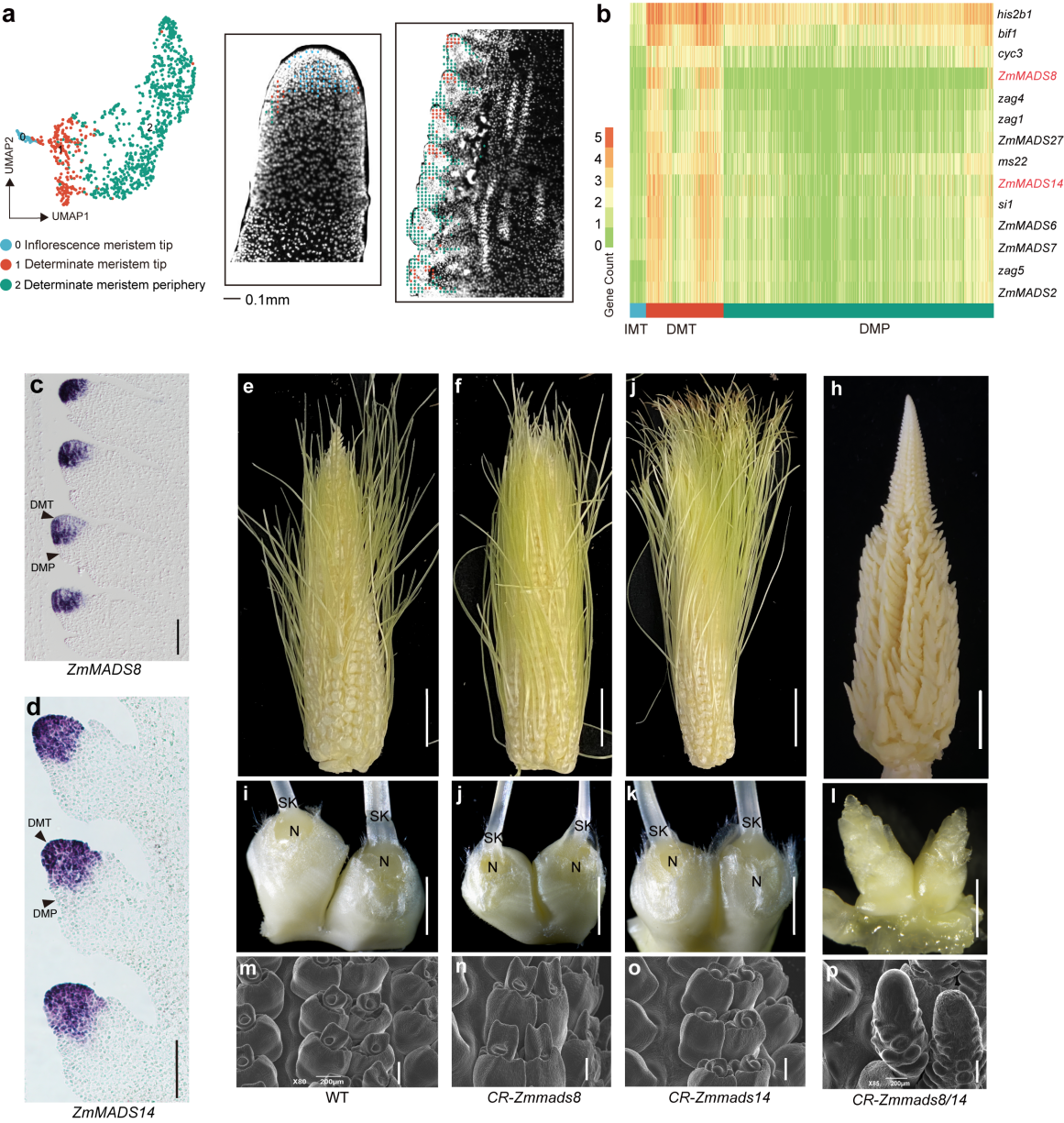
图2. 利用Stereo-seq数据鉴定到ZmMADS8和ZmMADS14决定小花分生组织确定性
接下来,研究人员利用STRIDE分析流程,将单细胞转录组与空间转录组数据进行了整合,通过空间映射,注释了单细胞转录组数据中的6种细胞类型,并根据不同细胞的转录组信息构建了具有空间表达特异性的基因调控网络。其中,研究人员在玉米雌穗分生组织的表皮细胞中鉴定到了以OCL5(OUTER CELL LAYER 5)和OCL3(OUTER CELL LAYER 3)基因为hub节点的共表达网络,这一共表达网络中还包含另外三种类型的基因,分别为3-酮酯酰-CoA合成酶基因(3-ketoacyl-CoA synthase)以及表皮蜡质合成基因(glossy genes)。OCL基因在形成籽粒表皮的角质层中发挥重要作用的结论已经在拟南芥以及玉米中被报道过,而在玉米雌穗的空间转录组图谱中,OCL5基因明确表现出在雌穗分生组织的表皮细胞中特异表达的特征。因此,研究人员推测在该部位存在一个以OCL基因为核心的,与角质层合成相关的基因调控网络,而这一结论与分生组织表皮细胞所行使的生物学功能高度吻合。此外,研究人员还鉴定到了一个在花序分生组织中特异性表达的共表达网络,该网络包含多个与海藻糖-6-磷酸合成相关的基因。而根据过去的研究,海藻糖-6-磷酸对于植物的生长发育非常重要。在该共表达网络中,另一个NAC家族的转录因子NACTF25被鉴定为hub基因,并且在花序分生组织中特异性表达。因此,研究人员推测NACTF25可能通过调控与海藻糖-6-磷酸合成相关的基因表达来影响花序分生组织发育(图3)。
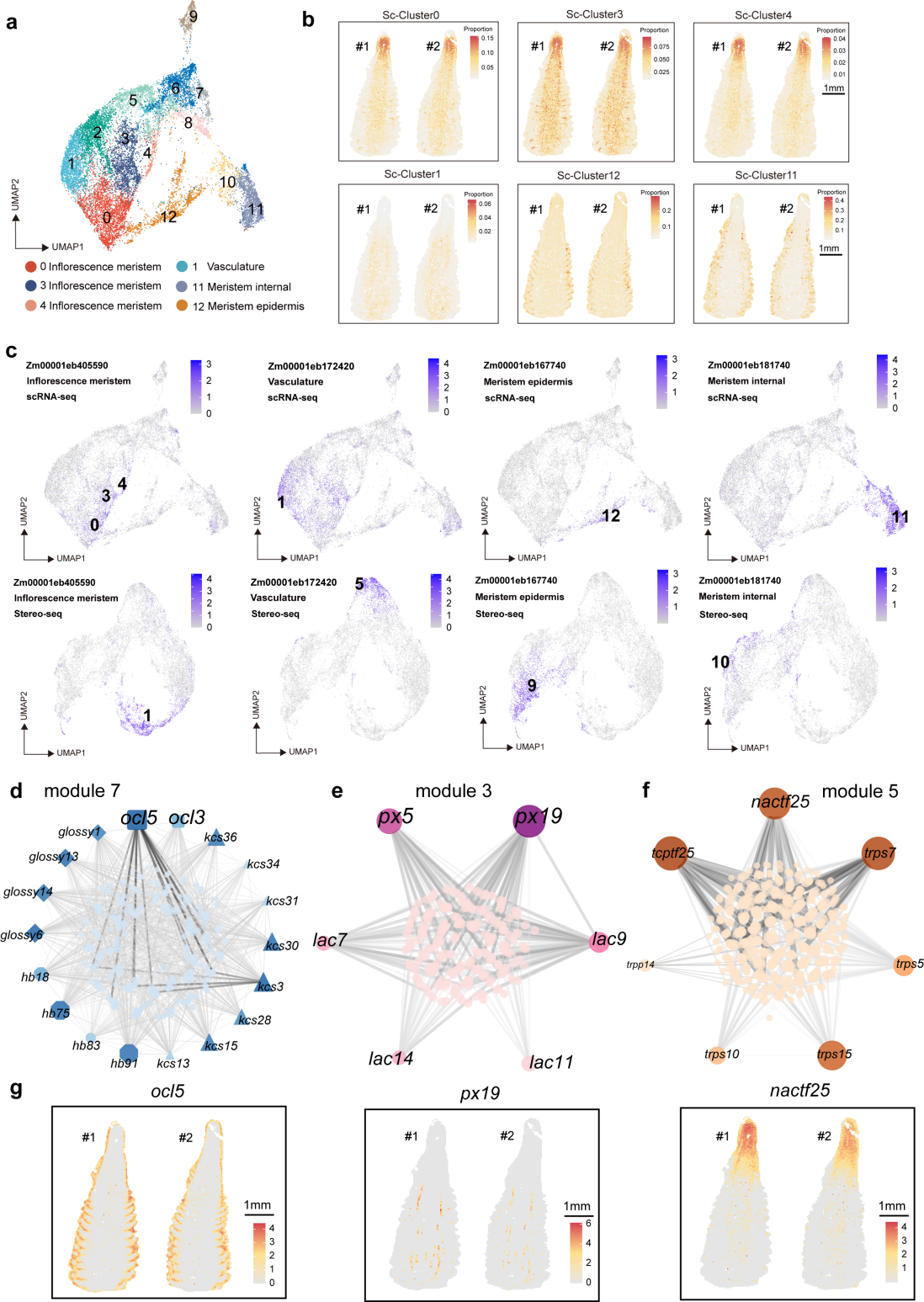
图3. 整合单细胞转录组与空间转录组数据构建细胞类型特异性共表达网络
综上所述,本研究通过结合关键发育时期下玉米雌穗的单细胞与空间转录组数据,构建了玉米雌穗的空间转录组图谱。利用该图谱能够区分高度相似的分生组织类型,使我们能够进一步研究分生组织在分化过程中产生的细微变化,并有助于鉴定关键的发育调控因子。图谱中所有基因的表达模式(表达量与表达部位)以及在不同细胞类型中鉴定到的标记基因信息均可以通过https://db.cngb.org/stomics/mdesta/访问查询。
华中农业大学作物遗传改良全国重点实验室和湖北洪山实验室杨宁教授、刘磊教授及深圳华大研究院刘欢研究员为该论文共同通讯作者。华中农业大学王跃斌博士、罗芸博士、李云富博士、严佳丽和深圳华大研究院郭兴博士为该论文的共同第一作者。华中农业大学严建兵教授、张祖新教授,美国冷泉港实验室David Jackson教授,深圳华大研究院徐讯研究员参与指导了该项工作。华中农业大学卓琳、魏文杰、丁倩、柏民基,以及深圳华大研究院魏晓锋、邵雯雯、陈丽华、黎丽、杨涛、陈静为研究提供了技术支持。该工作得到了农业生物育种重大项目、国家自然科学基金委优秀青年科学基金、国家重点研发计划等项目的资助。
审核人:杨宁
【英文摘要】
A comprehensive understanding of inflorescence development is crucial for crop genetic improvement, as inflorescence meristems give rise to reproductive organs and determine grain yield. However, dissecting inflorescence development at the cellular level has been challenging owing to a lack of specific marker genes to distinguish among cell types, particularly in different types of meristems that are vital for organ formation. In this study, we used spatial enhanced resolution omics-sequencing (Stereo-seq) to construct a precise spatial transcriptome map of the developing maize ear primordium, identifying 12 cell types, including 4 newly defined cell types found mainly in the inflorescence meristem. By extracting the meristem components for detailed clustering, we identified three subtypes of meristem and validated two MADS-box genes that were specifically expressed at the apex of determinate meristems and involved in stem cell determinacy. Furthermore, by integrating single-cell RNA transcriptomes, we identified a series of spatially specific networks and hub genes that may provide new insights into the formation of different tissues. In summary, this study provides a valuable resource for research on cereal inflorescence development, offering new clues for yield improvement.