南湖新闻网讯(通讯员 欧阳维枝)近日,我校作物遗传改良国家重点实验室李兴旺课题组研究成果以“Profiling Plant Histone Modification at Single-cell Resolution Using snCUT&Tag”为题在Plant Biotechnology Journal。研究首次报道了在植物中建立了单细胞核CUT&Tag (snCUT&Tag)方法,可以高通量分析单个细胞核的组蛋白修饰特征,本研究将开启植物单细胞表观基因组学研究的序幕。
在组织生长和细胞分化过程中,虽然细胞之间的基因组的序列是相同的,但是细胞之间存在表观遗传的异质性,细胞命运的决定方向也会有差异,最终导致细胞朝着不同的轨迹分化为不同的细胞类型。因此,研究细胞与细胞之间的组蛋白修饰等表观遗传的异质性和单细胞水平的基因转录调控,对理解细胞分化过程细胞命运决定以及基因转录的表观遗传调控的分子机理具有重要的意义。染色质免疫共沉淀结合测序技术(ChIP-seq)是分析组蛋白修饰和转录因子结合位点的方法。不过ChIP-seq和eChIP-seq等技术依赖于超声断裂染色质和免疫共沉淀等许多步骤,只能用大量细胞或者组织作为起始材料,其获得的是群体细胞的染色质特征的平均水平,并不能反映单个细胞中组蛋白修饰的真实状态 (Carter and Zhao, 2021, Nat Rev Genet)。染色质免疫切割结合测序技术(chromatin immunocleavage with sequencing, ChIC-seq)是基于抗体引导的protein A/G-Tn5融合蛋白在目标区域原位转座断裂染色质并同时带上标签序列的方法。ChIC-seq方法不依赖于超声断裂和免疫共沉淀等步骤,方法简单快速,可以实现分析单个细胞的染色质表观修饰特征,是ChIP-seq的很好的替代技术。目前,在动物细胞系中已经建立了很多单细胞ChIC-seq方法,包括scChIC-seq, CUT&Tag, CoBATCH和ACT-seq等等。但是,由于植物细胞壁等结构特性,目前还未见植物单细胞ChIC-seq方法的报道。
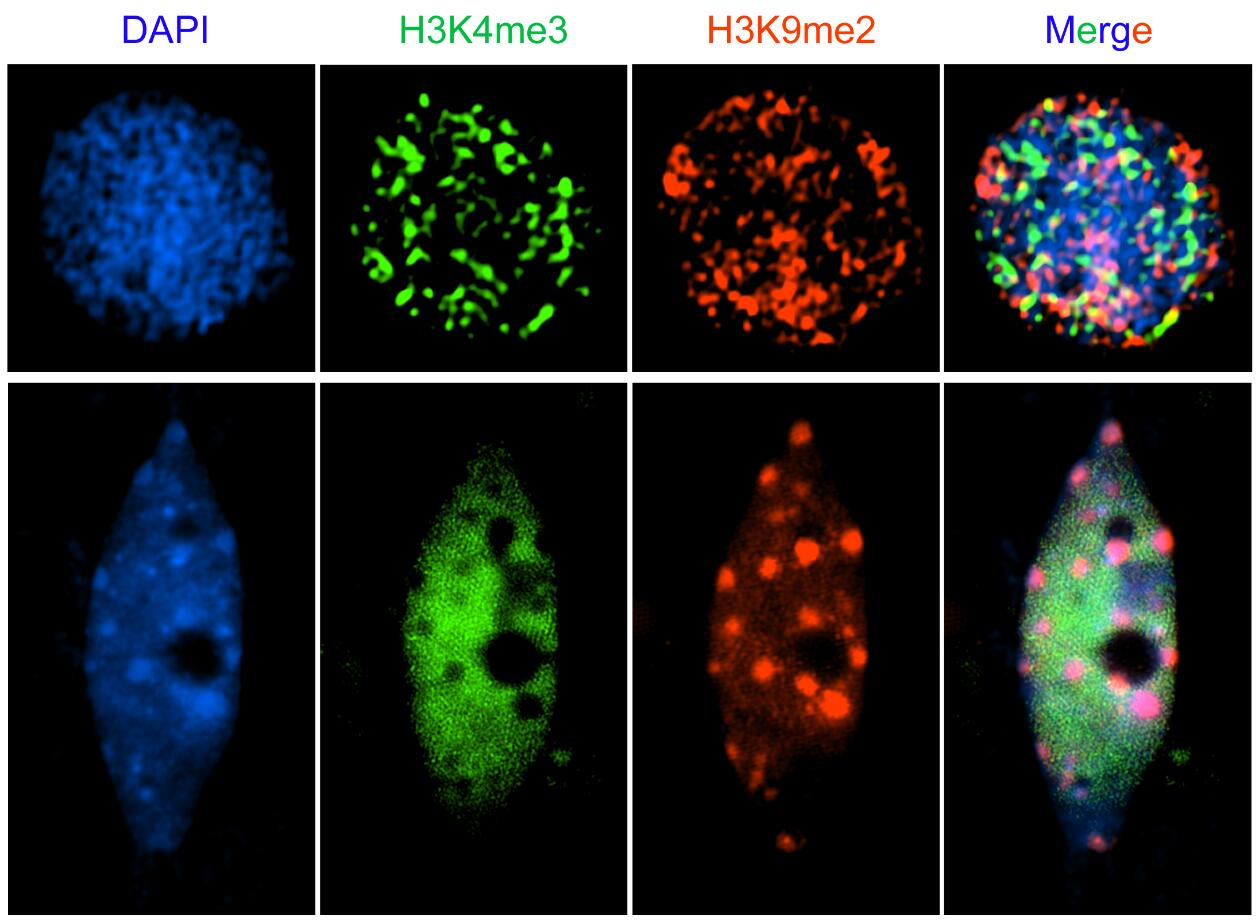
图1:细胞之间组蛋白修饰的异质性。上排为水稻幼苗中的细胞核,下图为油菜幼苗的细胞核
在前期研究中,李兴旺课题组建立了植物高效eChIP-seq技术 (Zhao et al., 2020, Nat Commun),并通过分离高质量的细胞核,在水稻和油菜等作物中建立了细胞核CUT&Tag (nCUT&Tag)技术,可以快速高效分析植物组蛋白修饰特征(Ouyang et al., 2021, Front Plant Sci)。在本研究中,作者进一步将nCUT&Tag技术与基于微流控的单细胞标记技术(10x Single Cell ATAC)相结合,首次建立了植物单细胞核CUT&Tag (snCUT&Tag)技术(如图2)。该方法首先分离水稻细胞核,随后将细胞核与抗体孵育,使抗体结合到靶蛋白上;然后与protein G-Tn5融合蛋白孵育,通过protein G与抗体的结合,将转座酶Tn5带到靶蛋白附近;加入Mg离子后,可以激活Tn5的转座活性,在靶蛋白附近原位切割DNA并在切口末端加上接头序列;Tn5切割并带上标签的细胞核加载到微流控单细胞标记芯片上,即可以标记并区分每一个单细胞的DNA信息,通过测序即可以分析成千上万个单个细胞的组蛋白修饰信息。
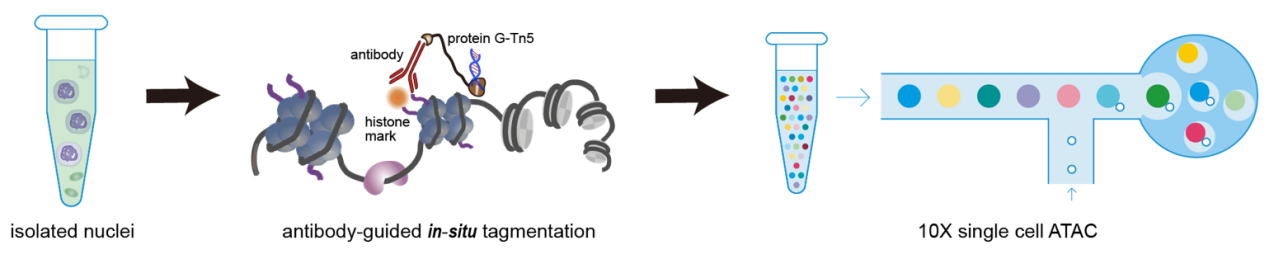
图2:snCUT&Tag的工作原理图
作者用snCUT&Tag分析了籼稻品种明恢63幼苗的单细胞组蛋白修饰H3K4me3的特征。本研究中一共捕获了3679个单细胞核的染色质特征,分析表明snCUT&Tag数据与相同组织的nCUT&Tag以及eChIP-seq数据有很好的相关性和一致性,H3K4me3的peak信号主要在转录起始位点富集,并且snCUT&Tag数据具有很好的信噪比。进一步分析不同细胞类型的表观遗传异质性发现,捕获的单细胞核聚类成17个cluster,对应的cluster具有很特异的marker peak信号,表明snCUT&Tag可以很好地分析细胞之间的表观遗传异质性(图3)。
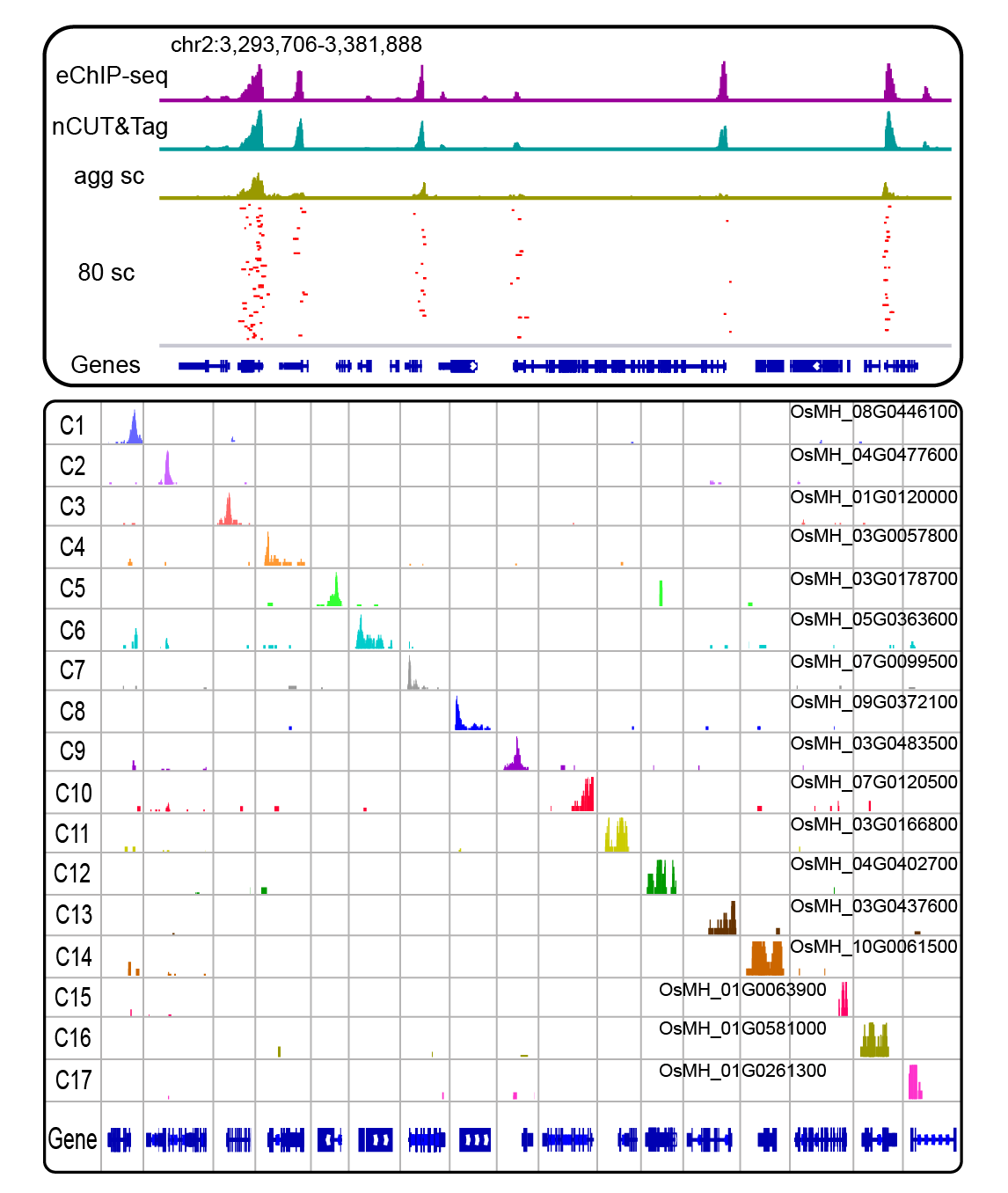
图3:单细胞核snCUT&Tag捕获的H3K4me3信号和marker peak的基因组截图展示
接下来,作者利用snCUT&Tag数据,依据ABC(activity-by-contact)模型预测了细胞类型特异的染色质互作。分析发现,snCUT&Tag预测的染色质互作与bulk cell的H3K4me3 ChIA-PET数据具有很好的一致性,并且预测的染色质互作的anchor与ATAC-seq的peak信号有很高的重合,暗示这些DNA序列可能发挥增强子的功能,并通过染色质互作调控相关基因的转录从而调控细胞分化命运以及其他生物学过程。这些结果为调控元件的注释、单细胞水平基因表达的表观遗传调控机理的解析以及单细胞核的三维基因组结构的重建提供了新的思路。
植物传统的ChIP-seq步骤繁多,所需的实验材料较多,实验时间较长,整体的效率偏低。李兴旺团队通过改进实验方法,建立了植物eChIP-seq技术,与传统植物ChIP-seq技术相比,eChIP-seq捕获染色质特征的效率提升了至少一百倍以上。利用eChIP-seq技术,课题组与合作者建立了水稻、油菜和玉米等作物的参考表观基因组(reference epigenome) (Zhao et al., 2020, Nat Commun; Zhang et al., 2021, Mol Plant; Sun et al., 2020, Genome Biol),并建立了水稻的DNA元件百科全书RiceENCODE (Xie et al., 2021, Mol Plant)。在eChIP-seq的基础上,课题组进一步建立了植物long-read ChIA-PET技术,绘制了水稻和玉米等作物的高分辨率三维基因组图谱(Zhao et al., 2019 Nat Commun; Peng et al., 2019 Nat Commun)。这些在植物表观基因组和三维基因组等领域取得的一系列进展,为深入理解作物基因转录调控机理提供了重要的方法和资源。
我校生科院博士后欧阳维枝与硕士研究生栾世平为文章共同第一作者。李兴旺教授为文章通讯作者,信息学院李国亮教授参与了课题指导。相关工作得到国家自然科学基金、我校交叉科学研究院、作物遗传改良国家重点实验室,以及生命科学技术学院“百川计划”的资助。
审核人:李兴旺
【英文摘要】
Chromatin immunoprecipitation with sequencing (ChIP-seq) with population cells or tissues yields ensemble epigenomic profiles that only represent the population average, which eliminates cell-to-cell epigenetic heterogeneity. Chromatin immunocleavage with sequencing (ChIC-seq) (Ku et al., 2019) that based on antibody-guided chromatin cleavage under targets is a practical alternative to ChIP-seq. Many single-cell ChIC-based methods, such as scChIC-seq (Ku et al., 2019), CUT&Tag (Kaya-Okur et al., 2019), CoBATCH (Wang et al., 2019), ACT-seq (Carter et al., 2019), and 10x scCUT&Tag (Bartosovic et al., 2021; Wu et al., 2021), have been developed and applied to study cell-type profiling, developmental trajectory, epigenetic heterogeneity, and transcriptional regulation in animals. However, due to the existence of cell walls, it is difficult to obtain single cells in plants. On the other hand, the existing scChIC methods require many single-cell barcoding procedures after tagmentation, which might lead to some DNA leakage and reduced mapping efficiency. Hence, a robust single-cell ChIC-seq method for plant epigenomic research is required emergently. In our previous study, we developed a rapid ChIC-based chromatin profiling protocol, nucleus CUT&Tag (nCUT&Tag) (Ouyang et al., 2021). Here, we combined the nCUT&Tag assay with 10x Single Cell ATAC, developing an easy-to-use single-nucleus CUT&Tag (snCUT&Tag) method in rice.
