南湖新闻网讯(通讯员 苏汉东)近日,华中农业大学植物科学技术学院小麦团队苏汉东教授课题组在《Genome Biology》杂志发表了“Three near-complete genome assemblies reveal substantial centromere dynamics from diploid to tetraploid in Brachypodium genus”的研究论文。该研究首次系统揭示了植物多倍化过程基因组最复杂区域着丝粒的遗传变异机制,为新物种形成和多倍体基因组的稳定提供新认知。

真核生物着丝粒是基因组结构最复杂、变化最剧烈的区域,富含高度相似的串联重复序列,中间穿插着长末端重复序列(LTR)转座子。着丝粒对于染色体的正确分裂和基因组稳定性的维持至关重要。着丝粒序列的快速变异可以促进核型进化和新物种形成。然而,物种间着丝粒的变异机制以及多倍体不同基因组来源的着丝粒在数百万年进化过程是如何协同稳定的,一直是领域内长期没有解决的重大生物学问题。
短柄草与麦类作物同属于早熟禾亚科,是小麦的近亲,因具有较小的基因组和较为成熟的实验系统,是研究小麦多倍化过程的模式系统。该研究利用短柄草属的异源四倍体杂交短柄草(Brachypodium hybridum,2n=4x=30)以及两个二倍体祖先二穗短柄草(B. distachyon,2n=2x=10)和斯特斯短柄草(B. stacei,2n=2x=20)。在单碱基水平系统解析了两个二倍体近缘种间着丝粒重复序列的组成、结构和表观遗传的巨大变异,表明来自共同起源的祖先种着丝粒重复序列在物种进化过程出现了分化,并且着丝粒内部的同质化伴随着局部卫星重复序列的迅猛增长,而反转座子的插入驱动了不同物种间着丝粒重复序列的多样性。
研究人员进一步研究了在异源多倍化过程着丝粒的动态变化,发现从二倍体到四倍体的过程中,着丝粒内部的结构重排驱动着丝粒遗传和表观遗传的快速转变。此外,泛基因组分析还揭示了短柄草属基因组个体间着丝粒重复序列发生持续变异。最后,研究人员通过和普通小麦异源多倍化过程亚基因组着丝粒的相互作用比较,提出了减数分裂早前期不同亚基因组着丝粒空间距离影响着丝粒变异的遗传机制。该研究为近缘物种及其异源多倍体基因组之间高度重复DNA序列的基因组学、表观基因组学和功能多样性方面提供了有力的见解,对于理解着丝粒种间分化驱动的新物种形成和多倍化核型稳定过程具有重要理论意义。
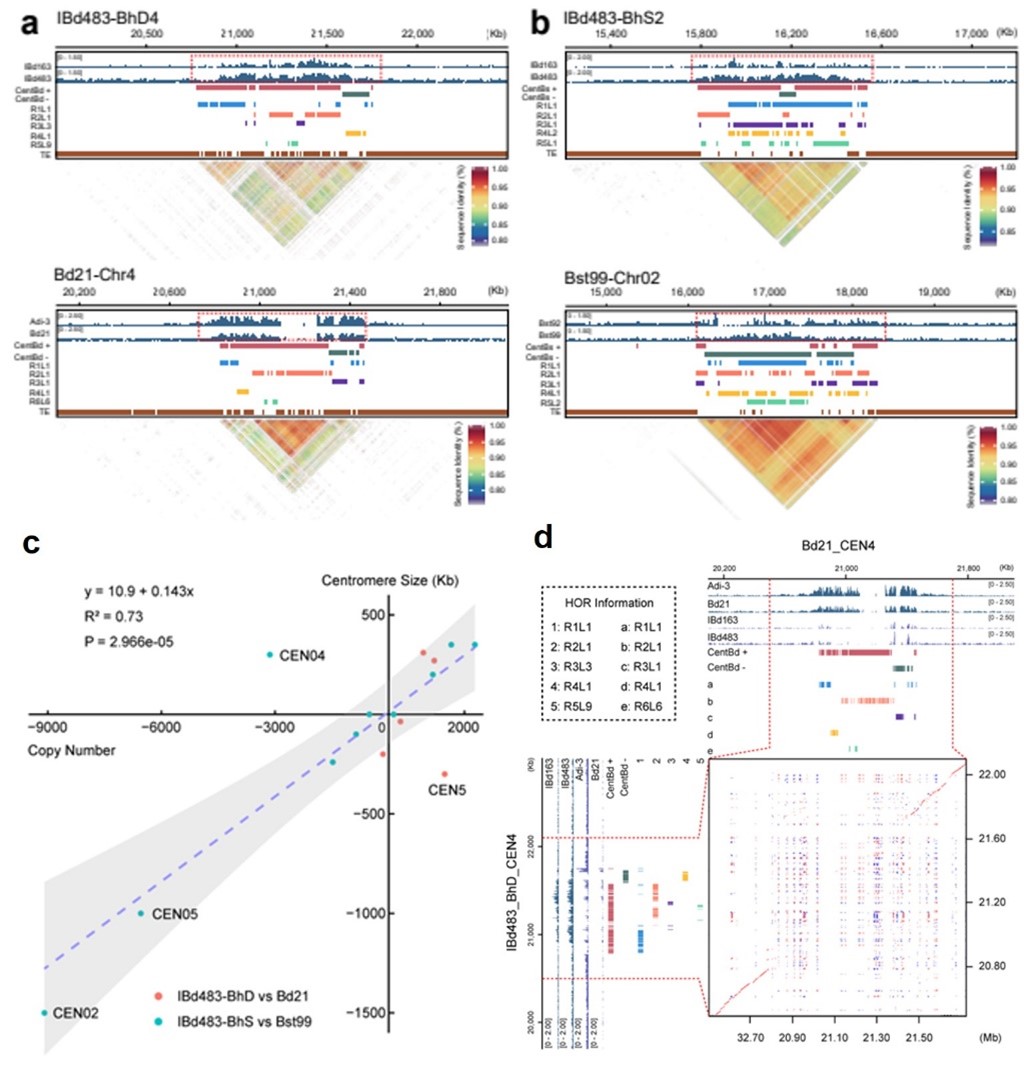
短柄草属不同物种着丝粒的序列、结构比较及多倍化过程动态变化
华中农大植科院博士研究生陈川晔、硕士研究生吴思颖和中科院遗传所孙翼双博士为论文共同第一作者,华中农大植科院小麦团队苏汉东教授为论文通讯作者,玉米团队杨宁教授、中科院遗传发育所韩方普研究员对该研究进行重要指导和建议。感谢中科院遗传发育所刘志勇研究员提供的遗传材料。该研究得到国家重点研发计划和国家自然科学基金等项目资助。
英文摘要:
Background
Centromeres are critical for maintaining genomic stability in eukaryotes, and their turnover shapes genome architectures and drives karyotype evolution. However, the co-evolution of centromeres from different species in allopolyploids over millions of years remains largely unknown.
Results
Here, we generate three near-complete genome assemblies, a tetraploid Brachypodium hybridum and its two diploid ancestors, Brachypodium distachyon and Brachypodium stacei. We detect high degrees of sequence, structural, and epigenetic variations of centromeres at base-pair resolution between closely related Brachypodium genomes, indicating the appearance and accumulation of species-specific centromere repeats from a common origin during evolution. We also find that centromere homogenization is accompanied by local satellite repeats bursting and retrotransposon purging, and the frequency of retrotransposon invasions drives the degree of interspecies centromere diversification. We further investigate the dynamics of centromeres during alloploidization process, and find that dramatic genetics and epigenetics architecture variations are associated with the turnover of centromeres between homologous chromosomal pairs from diploid to tetraploid. Additionally, our pangenomes analysis reveals the ongoing variations of satellite repeats and stable evolutionary homeostasis within centromeres among individuals of each Brachypodium genome with different polyploidy levels.
Conclusions
Our results provide unprecedented information on the genomic, epigenomic, and functional diversity of highly repetitive DNA between closely related species and their allopolyploid genomes at both coarse and fine scale.
审核人 鄢文豪