南湖新闻网讯(通讯员 芦慧)近日,我校生物医学与健康学院成锐教授与海洋三所海洋生物遗传资源重点实验室董西洋研究员团队合作,在国际学术期刊Nature Communications在线发表了题为“Co-occurrence of diverse defense systems shapes complex microbe-virus relationships in deep-sea cold seeps”的最新研究成果。该研究系统解析了冷泉生态系统中微生物防御系统与病毒抗防御系统的分布特征及互作关系,揭示了多类防御系统与抗防御系统在极端环境中形成的共演格局,为理解深部生命适应机制提供了新的研究基础。

深海冷泉是全球海洋中重要的极端生境,其高压、低温及富含碳氢化合物的环境孕育了多样的深部微生物群落。病毒在该环境中数量丰富、活动频繁,是影响微生物群落结构和生态功能的重要生物因子。
这项研究整合来自全球17个深海冷泉站位的191份沉积物样品的宏基因组和宏转录组数据,共获得3813个种水平的微生物基因组,以及13336个病毒基因组。随后在3813个微生物基因组中,鉴定出36783个防御相关基因和26389套防御系统,其中约63%为此前未见报道的候选防御系统,显示深海冷泉微生物具有丰富而多样的潜在抗病毒机制。
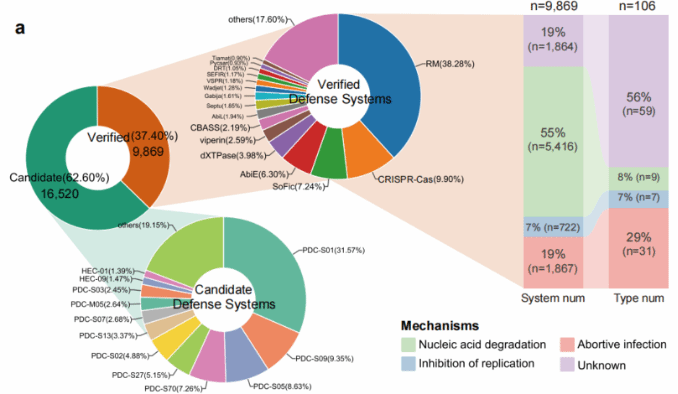
图1.冷泉微生物防御系统概况
多类防御系统在微生物基因组中表现出聚集特征,常以防御岛形式存在;相关性分析结果表明不同冷泉防御系统之间可能通过协同作用共同抵御病毒的侵染;结合宏转录组数据分析表明,这些防御系统在原位环境中具有一定的转录活性。
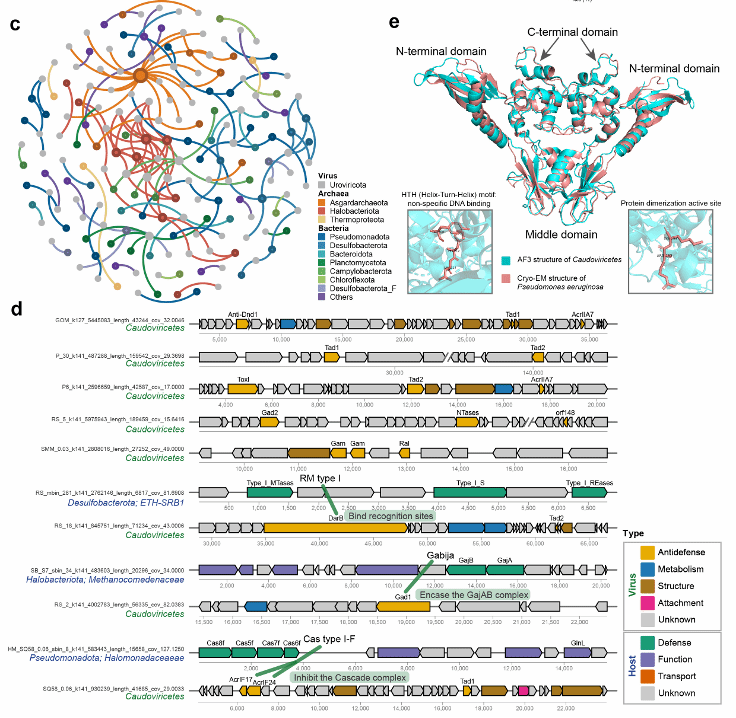
图2.冷泉微生物防御系统与病毒抗防御系统的互作
该研究还对部分候选防御系统开展了功能验证实验,多套系统在异源表达条件下表现出对不同噬菌体的抑制作用,表明深海冷泉环境中蕴含具有潜在应用价值的抗病毒分子资源,为理解极端生境中病毒—宿主互作机制提供了重要数据支撑。
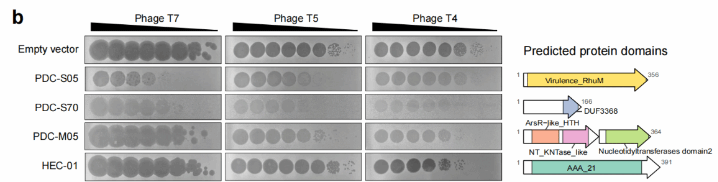
图3.冷泉候选防御系统的实验验证与结构域预测
这项研究为深海冷泉病毒与微生物在极端环境下的防御互作提供了系统证据。通过对大规模基因组数据的比较分析,该研究揭示了病毒压力在冷泉生态中的广泛影响,以及微生物通过多样化防御机制应对病毒侵染的适应策略。研究结果为深入认识深部生命的生态功能、环境响应机制,以及深海生物资源和新型生物防御机制的发掘提供了研究基础和科学依据。
海洋三所海洋生物遗传资源重点实验室董西洋研究员与我校生物医学与健康学院、生命科学技术学院成锐教授为共同通讯作者,海洋三所韩迎春助理研究员与硕士研究生廖静为共同第一作者,相关合作单位还包括浙江大学、中国科学院海洋研究所、大连海事大学、上海交通大学、宁波东方理工大学等。该研究得到国家重点研发计划和国家自然科学基金等资助支持。相关研究依托海洋三所海洋生物遗传资源重点实验室和我校农业微生物资源发掘与利用全国重点实验室等平台。
论文链接:https://www.nature.com/articles/s41467-025-68174-6
英文摘要:Cold seeps host diverse microbes and viruses with numerous unexplored defense and anti-defense systems. Analysis of 3813 microbial and 13,336 viral genomes from 191 metagenomes across 17 cold seep sites reveals extensive microbial defense repertoires, with over 60% representing candidate systems. Experimental validation confirms that several candidates protect against viral infection. These defense systems frequently co-occur, suggesting potential synergistic interactions, and are broadly distributed across sediments. In response, viruses have evolved diverse anti-defense genes, and the concurrent presence of multiple viral and microbial systems highlights intricate coevolution. Functionally critical lineages, such as anaerobic methanotrophic archaea, sulfate-reducing bacteria, and diazotrophs, appear to modify their defensive strategies under ecological and environmental pressures; for example, sulfate-reducing bacteria harbor multiple Gabija systems while corresponding viruses carry anti-Gabija genes, illustrating specific coevolutionary adaptations. Overall, these findings underscore the critical role of virus-microbe interactions in shaping microbial metabolic functions and environmental adaptation in deep-sea ecosystems.
审核人:成锐