南湖新闻网讯(通讯员 纪博明)盐碱地是重要的后备耕地资源。我国具有开发利用潜力的盐碱地约5亿亩。发掘玉米耐盐基因,解析玉米耐盐机制可为玉米耐盐育种提供理论指导和基因资源,具有重要的科学意义和实践价值。玉米盐胁迫应答是复杂的动态过程,系统了解玉米盐胁迫应答机制是进行玉米耐盐育种的前提。
2025年9月10日,我校作物遗传改良全国重点实验室代明球教授课题组联合以色列特拉维夫大学Yariv Brotman课题组在国际学术期刊 Genome Biology 发表了题为“High-resolution time-series transcriptomic and metabolomic profiling reveals the regulatory mechanism underlying salt tolerance in maize”的研究论文。该研究构建了玉米极端耐盐自交系HLZY和盐敏感骨干系JI853的高密度表达和代谢谱,开发了新的时序数据挖掘算法,阐明了两玉米自交系耐盐性差异的原因,并结合遗传和分子研究方法,鉴定并验证了多个新的玉米耐盐基因,为玉米耐盐育种提供了理论基础和改良靶点并为其它时间序列多组学研究提供了分析框架。
利用RNA-seq, GC-MS和LC-MS技术,研究者获取了玉米极端耐盐自交系HLZY和敏感骨干系JI853在盐胁迫和对照下连续14个时间点的转录和代谢变化。利用偏最小二乘-判别(PLS-DA),似然比检验,年谱分析等多种数据分析手段,研究者发现耐盐自交系对盐胁迫存在更广泛和更迅速的转录和代谢应答。几丁质代谢与应答,肌醇和脯氨酸代谢,ABA与乙烯等激素应答等逆境响应过程在耐盐自交系中更早应答。利用时间序列聚类技术,研究者发现多个已知耐盐基因在耐盐自交系中表现出“提前激活”的表达模式,研究者验证了具有相同表达模式,但此前未报道参与玉米盐胁迫应答的新基因ZmGLK44是玉米盐胁迫应答的正调控因子。此外,研究者提出了一种基于LASSO的决策算法,整合了转录和代谢组数据,构建了玉米耐盐关键代谢物的基因调控网络,并验证了代谢-调控网络中的枢纽基因ZmGLN2的代谢调控功能。高达80%的ZmGLN2预测关联代谢物在zmgln2突变体中发生显著含量变化。zmgln2突变体的代谢组分析显示该基因缺失在盐胁迫下特异地抑制了玉米的氨基酸积累。
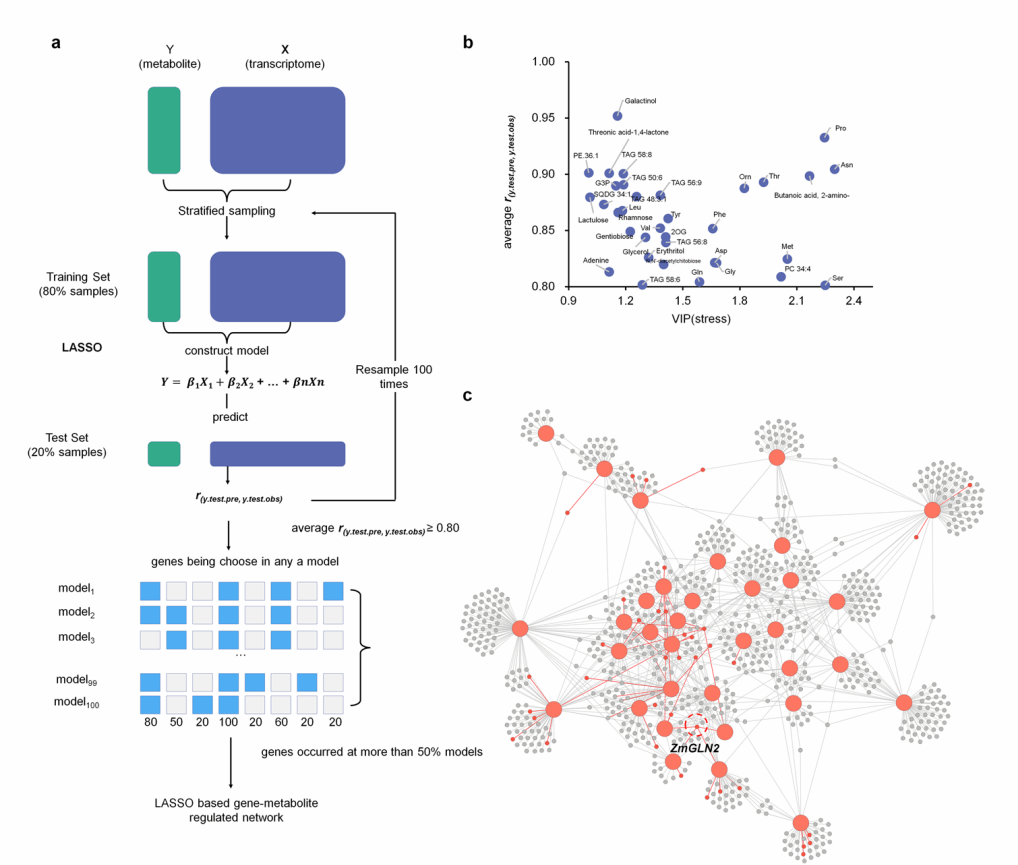
本研究基于耐盐性不同的玉米自交系,利用多种分析方法,阐明了玉米盐胁迫应答的关键过程,发现了新的玉米耐盐基因和代谢物,为玉米耐盐育种提供了基因资源,并为时序数据挖掘提供了新的分析框架。
华中农业大学已出站博士后,现北京大学现代农业研究院副研究员张斐,华中农业大学在读博士生纪博明,斯坦福大学吴思博士为论文的共同第一作者。华中农业大学代明球教授、以色列特拉维夫大学Yariv Brotman教授为本文的通讯作者。华中农业大学博士后张卉、博士生张杰、硕士生王飞参与了本研究。西北农林科技大学宋宝兴教授、广州大学桑青教授、广东农科院晏石娟研究员、黄文洁副研究员、德国马克思普朗克研究所分子植物生理所Mustafa Bulut博士对该研究提供了指导和帮助。该研究得到国家自然科学基金、中国博士后科学基金、湖北省自然科学基金、以及华中农业大学生命科学技术学院百川计划的资助。
英文摘要:
Background
Soil salinization represents a critical global challenge to agricultural productivity, profoundly impacting crop yields and threatening food security. Plant salt-responsive is complex and dynamic, making it challenging to fully elucidate salt tolerance mechanism and leading to gaps in our understanding of how plants adapt to and mitigate salt stress.
Results
Here, we conduct high-resolution time-series transcriptomic and metabolomic profiling of the extremely salt-tolerant maize inbred line, HLZY, and the salt-sensitive elite line, JI853. Utilizing advanced data mining techniques, we identify key factors underlying the divergence in salt tolerance between these two lines and discover a series of novel genes and metabolites essential for maize salt tolerance. Additionally, we develop an innovative decision algorithm that enabled the construction of a high-confidence gene regulatory network for important salt-responsive metabolites. Comprehensive genetic and molecular studies further reveal the pivotal role of a hub gene, ZmGLN2, in regulating metabolite biosynthesis and salt tolerance in maize.
Conclusions
Our study provides the first high-resolution transcriptomic and metabolomic dataset for crop salt response, uncovering novel maize salt-responsive genes and metabolites. These findings demonstrate the effectiveness of high-resolution multi-omics in deciphering the mechanisms underlying complex crop traits. Furthermore, we develop a systematic analytical framework for mining time-series multi-omics data, which can be broadly applied to other species or traits.
论文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-025-03766-5
审核人:代明球