南湖新闻网讯(通讯员 任学明)近日,植物科学技术学院牛长缨教授团队在BMC BIOLOGY上发表了最新成果"Gut symbiotic bacteria are involved in nitrogen recycling in the tephritid fruit fly Bactrocera dorsalis"。该文以世界性重大入侵害虫——橘小实蝇Bactrocera dorsalis为对象,从生理生化、分子水平系统阐释了肠道共生菌介导的橘小实蝇氮营养获取策略,筛选出介导幼虫氮营养获取途径的优势共生菌,揭示了共生菌介导的橘小实蝇氮循环的调控机制,为宿主昆虫——共生菌营养互作模式提供了新的认知。
植食性昆虫的取食、发育及繁殖取决于植物营养。对昆虫而言,通常其寄主植物中碳氮比高,可利用氮营养匮乏,同时大量氮被昆虫以含氮代谢废物的形式排出体外,因此氮营养胁迫是限制植食性昆虫生长发育的关键因素。
该研究对橘小实蝇成虫、幼虫、蛹及13株实蝇肠道潜在固氮菌进行固氮酶活测定,表明仅产酸克雷伯菌Klebsiella oxytoca具有固氮酶活性,其他样品并未表达出固氮酶活力,暗示在自然条件下固氮菌不能稳定地通过生物固氮为宿主提供氮营养。另一方面,发现随着果实内幼虫的发育,虫果中尿素含量逐渐积累;利用15N稳定同位素示踪法研究了橘小实蝇对尿素氮的同化、吸收能力,结果显示使用15N标记的尿素饲喂成虫、幼虫后,其体内不同组织中的δ15N及15N%值均显著提高,抗生素处理后的橘小实蝇组织内δ15N及15N%值显著低于未经抗生素处理组的相应δ15N及15N%值,表明肠道共生菌可将尿素氮转化为实蝇可利用的氮营养,提高其组织内氮含量。
利用宏基因组及宏转录组测序技术,分析了生物固氮与尿素水解通路中关键基因在共生菌基因组中的表达情况,表明固氮酶编码基因 (nifH、nifK) 转录缺失,导致固氮酶合成受阻,阻断了电子传递过程,因此氮不能被肠道共生菌固定。另一方面,编码脲酶复合体的URE、ureA、ureB、ureC、ureAB基因被成功转录,进而催化尿素水解产生氨。利用选择培养基从橘小实蝇肠道挖掘了Morganella morganii、Citrobacter freundii、K. oxytoca、K. pneumonia四株潜在尿素水解菌,测定了其生长曲线、脲酶活力及脲酶基因表达量,结果显示M. morganii在酸性条件下脲酶活性更强,另外三株细菌更适合在中性条件下表达脲酶活力,表明上述优势菌在橘小实蝇同化尿素氮的过程中发挥了关键作用。
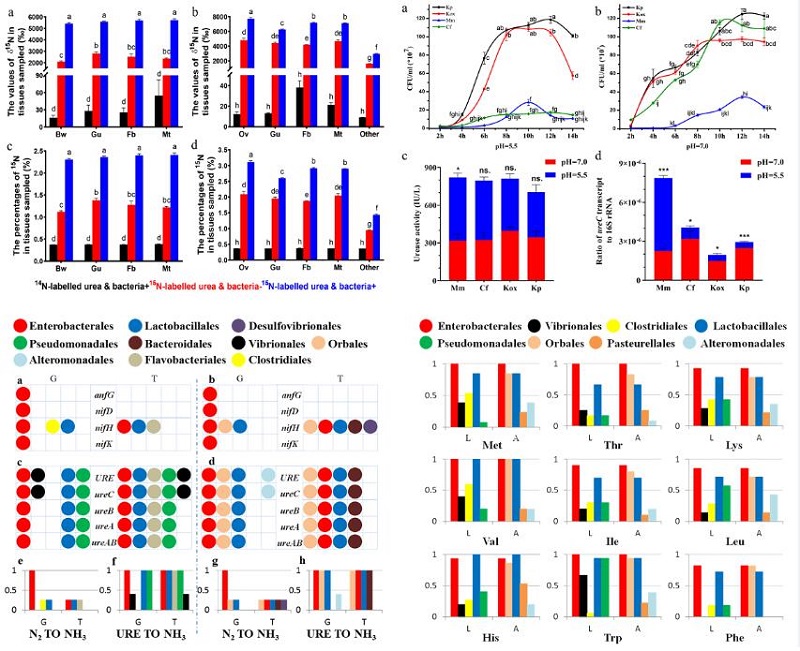
进一步构建了共生菌介导的含氮代谢废物降解及必需氨基酸合成通路汇总图。其中,含氮代谢废物如嘌呤、尿酸、尿囊素、尿囊酸、尿素等经系列生物酶催化后水解为氨;氨被谷氨酸脱氢酶或氧化还原酶催化合成谷氨酸 (GLU) 或天冬氨酸 (ASP);GLU及ASP在氨基转移酶的催化下作为氨基供体参与必需氨基酸的合成。
在共生菌介导的必需氨基酸合成通路中,共涉及115个功能基因,匹配结果显示介导精氨酸合成的argF基因在宏基因组注释结果中缺失,其余功能基因均存在于橘小实蝇成虫及幼虫肠道共生菌基因组中,说明其肠道共生菌具备合成必需氨基酸(精氨酸除外)的能力。从共生菌在必需氨基酸合成过程中的参与程度上分析,Enterobacterales和Lactobacillales目细菌主要介导幼虫必需氨基酸的合成;Enterobacterales、Orbales和Lactobacillales目细菌主要介导成虫必需氨基酸的合成。
华中农业大学在读博士研究生任学明为论文第一作者,牛长缨教授为论文通讯作者,该研究是由国家自然科学基金国际(地区)合作与交流项目 (31661143045)和国家自然科学基金面上项目 (31972270) 资助。
审核人:牛长缨
【英文摘要】
Background
Nitrogen is considered the most limiting nutrient element for herbivorous insects. To alleviate nitrogen limitation, insects have evolved various symbiotically mediated strategies that enable them to colonize nitrogen-poor habitats or exploit nitrogen-poor diets. In frugivorous tephritid larvae developing in fruit pulp under nitrogen stress, it remains largely unknown how nitrogen is obtained and larval development is completed.
Results
In this study, we used metagenomics and metatranscriptomics sequencing technologies as well as in vitro verification tests to uncover the mechanism underlying the nitrogen exploitation in the larvae of Bactrocera dorsalis. Our results showed that nitrogenous waste recycling (NWR) could be successfully driven by symbiotic bacteria, including Enterobacterales, Lactobacillales, Orbales, Pseudomonadales, Flavobacteriales, and Bacteroidales. In this process, urea hydrolysis in the larval gut was mainly mediated by Morganella morganii and Klebsiella oxytoca. In addition, core bacteria mediated essential amino acid (arginine excluded) biosynthesis by ammonium assimilation and transamination.
Conclusions
Symbiotic bacteria contribute to nitrogen transformation in the larvae of B. dorsalis in fruit pulp. Our findings suggest that the pattern of NWR is more likely to be applied by B. dorsalis, and M. morganii, K. oxytoca, and other urease-positive strains play vital roles in hydrolysing nitrogenous waste and providing metabolizable nitrogen for B. dorsalis.
论文链接:https://bmcbiol.biomedcentral.com/articles/10.1186/s12915-022-01399-9#Abs1