南湖新闻网讯(通讯员 张维奕)近日,我校果蔬园艺作物种质创新与利用全国重点实验室、园艺林学学院茶学团队闻玮玮教授课题组在Nature Protocols发表题为“Plant sperm cell sequencing for genome phasing and determination of meiotic crossover points”的研究论文。该论文详细介绍了一种依托单精子测序技术的基因组分型新方法。

单倍型信息在基因组学和遗传研究中起到了至关重要的作用,比如在填补缺失基因型数据、研究重组交换模式以及追溯物种演化历史等方面的应用,其对单体型基因组的组装也具有重要价值。然而,对于复杂基因组的单倍型分型仍是一个亟待解决的重大挑战。为攻克这一难题,该研究团队以茶树为实验材料,利用配子体这一天然的单倍体,基于单细胞测序技术,开发了一套全面且系统的流程。该流程由28个步骤构成,包括显微操作以及基因组扩增和测序等“湿实验”和生物信息学分析,涵盖了植物单精子的分离、单倍型分型以及重组交换位点的检测(图1)。这套方案能够精确地识别单精子间单核苷酸多态性(SNP),并基于此构建高密度的遗传图谱,从而可助于实现精准的染色体级别分型,有效地检测重组事件,并可应用于纠正潜在的基因组组装错误。生物信息分析流程可在Linux系统上高效运行,通过Docker镜像的方式,极大地简化了分析流程,降低了操作难度。这一创新不仅提升了分析效率,使得整个工作流程在一天内即可完成,而且展现了其在基因组学等研究中的巨大潜力和应用价值,为研究人员提供了更为便捷、高效的工具,助力其在基因组学等生物学研究领域取得更为深入的突破。
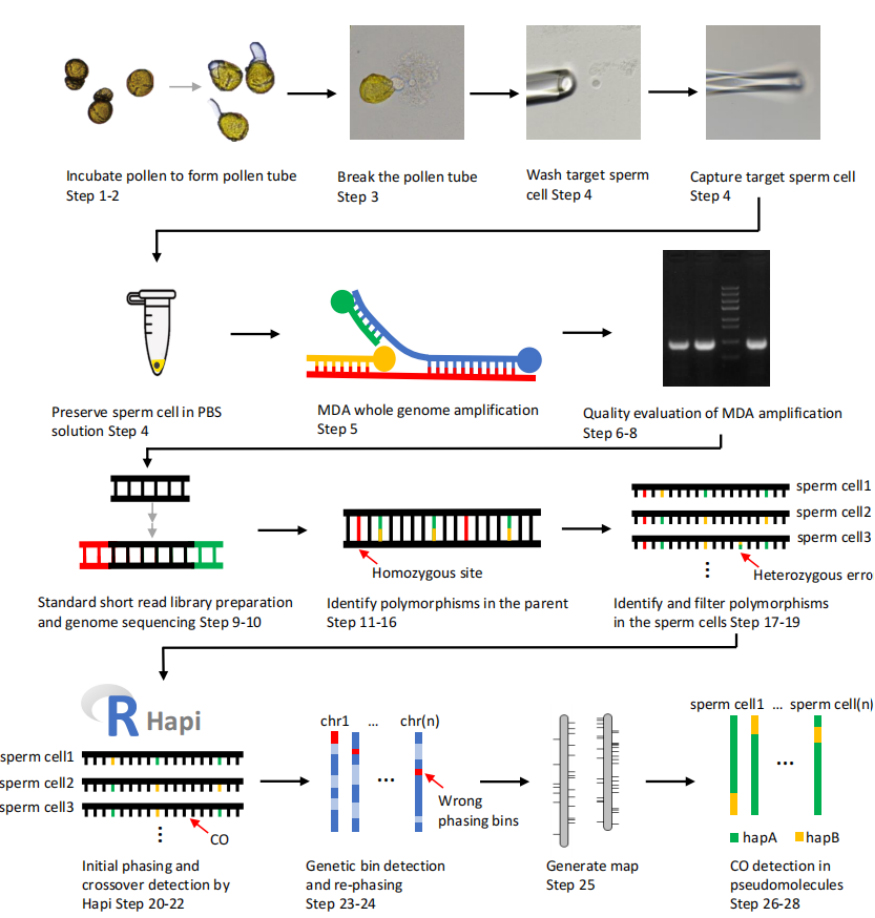
图1. 流程概况
果蔬园艺作物种质创新与利用全国重点实验室,湖北洪山实验室,华中农业大学园艺林学学院博士后张维奕、博士生贾欣欣和德国杜塞尔多夫大学博士生Arslan Tariq为论文的并列第一作者。果蔬园艺作物种质创新与利用全国重点实验室,湖北洪山实验室,华中农大园艺林学学院闻玮玮教授,德国杜塞尔多夫大学Björn Usadel教授和马克斯普朗克分子植物生理所Alisdair Fernie教授为共同通讯作者,华中农大严建兵教授参与了该项目研究。
审核人 闻玮玮
【英文摘要】
Haplotype phasing represents a pivotal procedure in genome analysis, entailing the identification of specific genetic variant combinations on each chromosome. Achieving chromosome-level genome phasing constitutes a considerable challenge, particularly in organisms with large and complex genomes. To address this challenge, we have developed a robust, gamete cell-based phasing pipeline, including wet-laboratory processes for plant sperm cell isolation, short-read sequencing and a bioinformatics workflow to generate chromosome-level phasing. The bioinformatics workflow is applicable for both plant and other sperm cells, for example, those of mammals. Our pipeline ensures high-quality single-nucleotide polymorphism (SNP) calling for each sperm cell and the subsequent construction of a high-density genetic map. The genetic map facilitates accurate chromosome-level genome phasing, enables crossover event detection and could be used to correct potential assembly errors. Our bioinformatics pipeline runs on a Linux system and most of its steps can be executed in parallel, expediting the analysis process. The entire workflow can be performed over the course of 1 d. We provide a practical example from our previous research using this protocol and provide the whole bioinformatics pipeline as a Docker image to ensure its easy adaptability to other studies.
点击阅读论文