南湖新闻网讯(通讯员 黄鲜晖)近日,我校棉花遗传改良团队在Nature Communications在线发表了题为Epigenomic and 3D genomic mapping reveals developmental dynamics and subgenomic asymmetry of transcriptional regulatory architecture in allotetraploid cotton的研究论文。该研究开启了棉花DNA元素百科全书(ENCODE)计划,揭示了亚基因组三维空间结构和表观修饰状态在全生育期中的动态重塑特征,解析了亚基因组表达不对称的表观调控机制,探究了非编码功能元件在纤维品质改良过程中的遗传调控效应及其育种价值。
亚基因组分歧与异源多倍体植物重要性状形成和环境适应性相关。亚基因组表达不对称可能反映了亚组同源基因的功能分化,在细胞命运决定、发育和免疫等诸多生物学过程都能检测到。异源四倍体陆地棉是由A和D基因组的两个二倍体祖先通过异源加倍形成,是当前全球种植面积最广的棉花栽培种,贡献95%以上纤维产量。华中农业大学棉花遗传改良团队长期致力于研究异源四倍体棉花亚基因组的演化分歧和对重要性状的协同调控机制。近三年,该团队解析了两个亚基因组动态协同调控纤维发育和品质形成的遗传机制,揭示了不同倍性棉花栽培种纤维品质平行育种选择的遗传共性和分歧,为从亚基因组水平开展优质棉花设计育种提供了理论支撑。
近年来,棉花功能基因研究和基因编辑技术发展非常迅速,为了全面破解棉花基因组序列的功能,在后基因组学时代开启棉花ENCODE计划意义重大。为了深化对棉花功能基因组的理解,解锁性状改良潜力,该研究以染色质三维结构、全局表观基因组状态和修饰为基础,对亚基因组同源基因在全生育期12个组织/发育阶段的转录偏向性变化和动态调控机制进行全面、深刻的解析。研究发现,陆地棉中约70%的亚基因组同源基因可表现出亚基因组偏向性表达,其中组织优势表达基因的偏向性表达程度更为显著,暗示组织特异生物学过程可能面临更突出的选择压力。
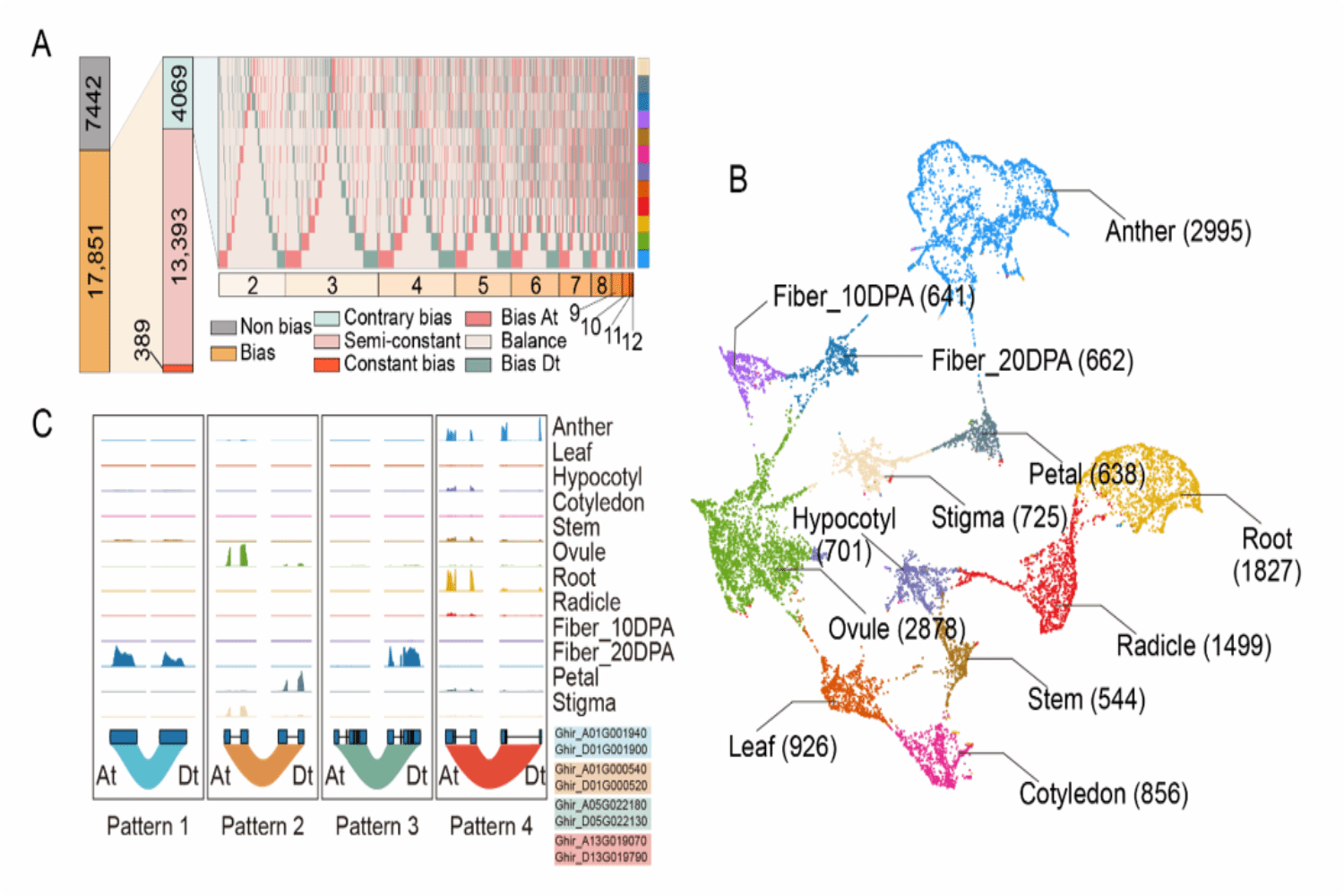
图1. 棉花全生育期同源基因偏向性/组织优势表达图谱
为探究棉花全生育期亚组同源基因偏向性表达的表观调控机制,该研究深度解析了棉花多组织的亚基因组多层级特征结构(从Mb到kb)特征。研究发现Dt亚基因组具有更频繁的组织间A/B compartment状态转换 (23.29% vs 15.30%),反映出Dt亚组包含更广泛的组织特征区间。此外,该研究创造性地提出了组织特异高活性TAD (TS-TADs),此类特征结构不仅表现出高转录活性,同时伴随高度活跃的sub-compartments状态,反映出染色质结构域呈现组织功能化的趋势。该研究发现~85%的TAD-like domains 边界在组织间保守,亚组间同源TAD-like domains 重排改变了~50%的同源偏向性基因对在TAD-like domains内部的相对位置,而其中约一半的基因位于TAD-like domains边界且偏向该亚组表达,说明亚基因组TAD-like domains重排可能是亚组同源基因表达分歧的重要因素。
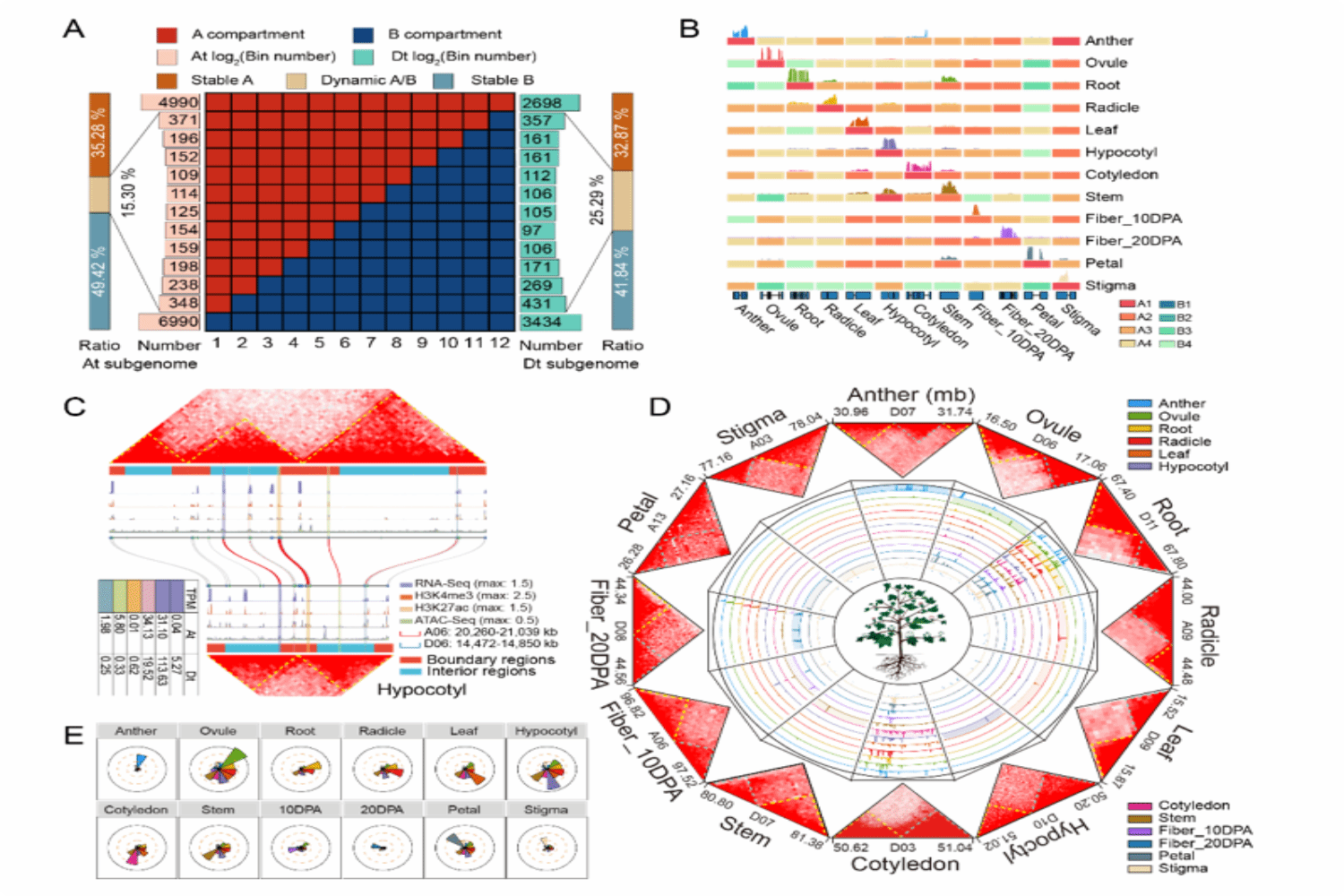
图2. 棉花亚基因组多层级三维空间结构
该研究开发了预测ATAC-Seq数据支持的组织调控元件鉴定方法(PATREs),以染色质可及性区间为基础,进一步结合组蛋白修饰信号,用于预测棉花非编码区潜在功能元件。研究发现31.63%的潜在功能调控元件(CRE)表现出亚基因组同源性,同源基因启动子区域染色质开放性或转录因子结合基序差异都将影响同源基因偏向性表达。分析精细染色质空间结构染色质环(loop)发现loop长度、染色质非编码区活性和loop多步调控方式都具备影响靶基因转录活性的潜力。比较亚基因组loop网络发现亚组间同源CRE缺失、loop调控缺失和loop位点改变将影响同源基因偏向性表达。以上结果说明,亚组三维空间重排和染色质表观修饰状态差异是造成不同组织同源基因间受到非一致性调控的关键因素。
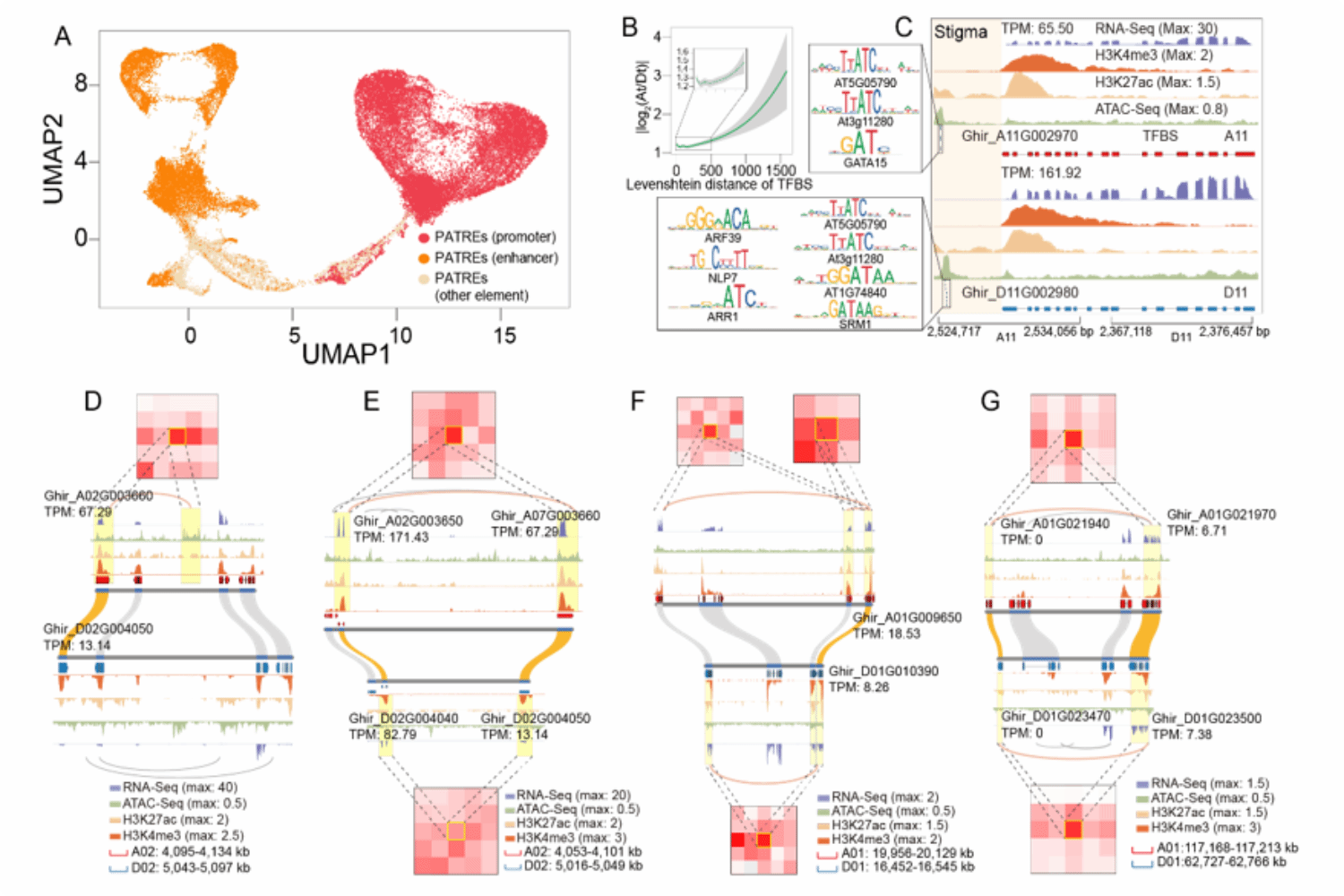
图3. 棉花亚基因组表观调控分歧
结合陆地棉群体纤维动态发育 (开花后4天 (4 DPA), 8 DPA, 12 DPA, 16 DPA, 20 DPA) 转录组,该研究进一步评估了CRE用于聚合育种的潜力。基于CNV-QTL和SNP-QTL,探索了CRE数量和序列变异与纤维品质性状改良间的关联性。该研究发现7个CREs拷贝数增加正向推动纤维品质改良,其中包含一个同时调控多个性状的多效性CRE。基于单倍型分析发现有18个纤维相关的CREs尚未被育种充分利用,存在良好的聚合育种潜力。结合CREs遗传变异以及CREs与靶基因间的三维调控关系,推测CREs变异可能引起关键染色质三维调控关系重排,进而影响潜在候选基因对性状改良的驱动力。最后,该研究开发了可用于比较、检索、可视化等多功能的棉花ENCODE网站 (http://cotten.hzau.edu.cn/),推动了棉花基因组的深度解析和功能基因组学的发展。
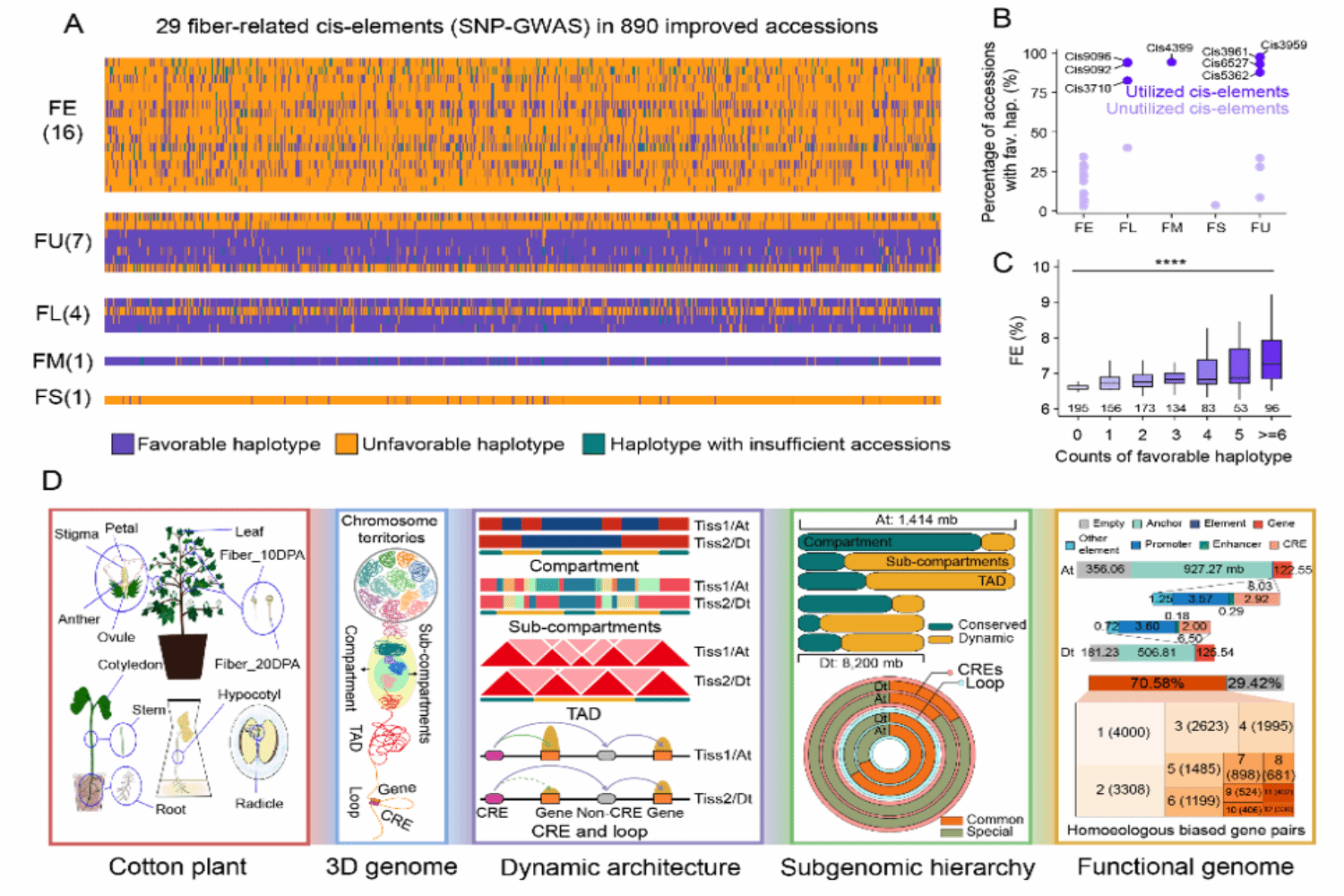
图4. CRE驱动棉花纤维品质改良
华中农业大学作物遗传改良全国重点实验室博士后黄鲜晖、博士生王悦瑾和研究生张赛男为该论文的共同第一作者,王茂军教授为通讯作者,张献龙院士和朱龙付教授参与了该项研究。该研究得到了国家自然科学基金、国家重点研发计划等项目资助。
【英文摘要】
Although epigenetic modification has long been recognized as a vital force influencing gene regulation in plants, the dynamics of chromatin structure implicated in the intertwined transcriptional regulation of duplicated genes in polyploids have yet to be understood. Here, we document the dynamic organization of chromatin structure in two subgenomes of allotetraploid cotton (Gossypium hirsutum) by generating 3D genomic, epigenomic and transcriptomic datasets from 12 major tissues/developmental stages covering the life cycle. We systematically identify a subset of genes that are closely associated with specific tissue functions. Interestingly, these genes exhibit not only higher tissue specificity but also a more pronounced homoeologous bias. We comprehensively elucidate the intricate process of subgenomic collaboration and divergence across various tissues. A comparison among subgenomes in the 12 tissues reveals widespread differences in the reorganization of 3D genome structures, with the Dt subgenome exhibiting a higher extent of dynamic chromatin status than the At subgenome. Moreover, we construct a comprehensive atlas of putative functional genome elements and discover that 37 cis-regulatory elements (CREs) have selection signals acquired during domestication and improvement. These data and analyses are publicly available to the research community through a web portal. In summary, this study provides abundant resources and depicts the regulatory architecture of the genome, which thereby facilitates the understanding of biological processes and guides cotton breeding.
点击阅读原文
审核人 王茂军