南湖新闻网讯(通讯员 陈鑫媛)近日,我校棉花遗传改良团队在Advanced Science在线发表了题为“Genetic regulatory perturbation of gene expression impacted by genomic introgression in fiber development of allotetraploid cotton”的文章,研究了遗传渐渗引起的背景基因组基因表达调控扰动,并揭示了其在农艺性状形成中的重要作用。同时,该研究从变异、调控和多倍化的维度讨论了基因渐渗在表型创新中的重要性,为优质棉纤维定向育种提供了丰富的遗传资源。
棉花(Gossypium spp.)是纺织工业中最重要的天然纤维来源。其中,陆地棉的纤维产量高、适应性广,在全世界内广泛种植;海岛棉的纤维品质优异,但纤维产量较陆地棉低。该研究以海岛棉(Gossypium barbadense,3-79)为供体,陆地棉(Gossypium hirsutum,Emian22)为受体的渐渗系群体为基础,揭示了伴随纤维品质变化的基因表达调控扰动现象(图1)。
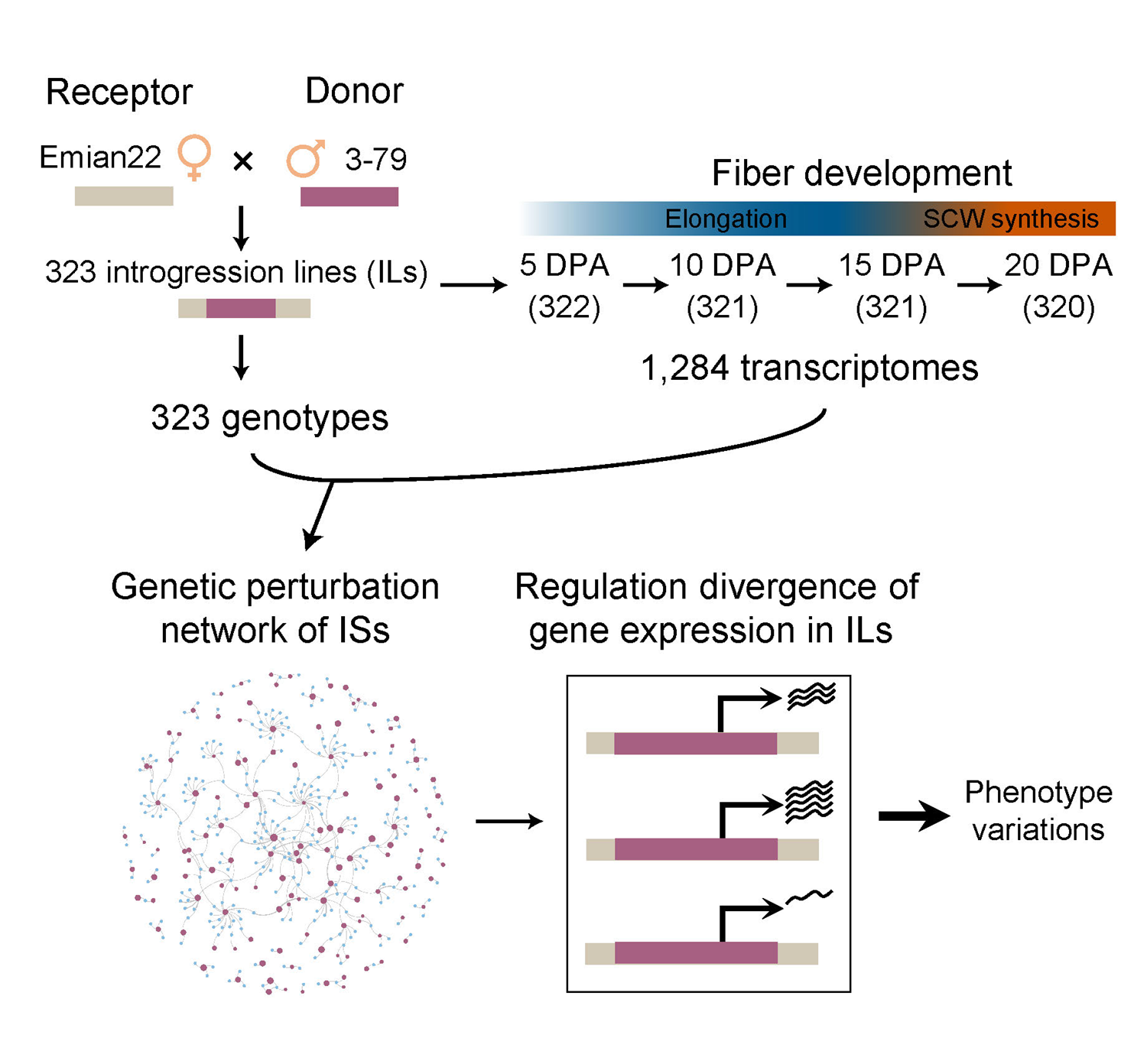
图1. 研究工作的模式图
研究从头组装了受体Emain22的基因组,获得了长度为2.24 Gb的基因组序列。通过比较陆地棉Emian22和海岛棉3-79的基因组序列,构建了两个棉种间的变异图谱,为解析渐渗系群体中的遗传信息提供了指导。随后以Emian22为参考基因组,在323份渐渗系中鉴定到1,217条来自海岛棉3-79的渐渗片段,覆盖了约85.43%的Emian22基因组序列(图2)。
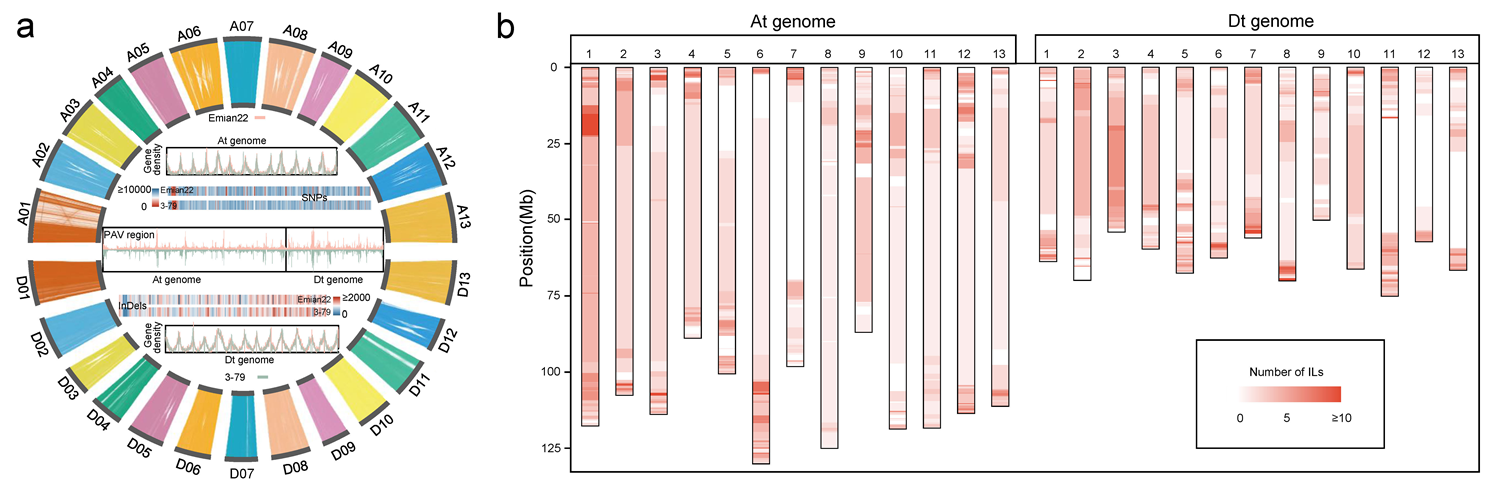
图2. Emian22基因组的渐渗变异图谱
结合纤维发育4个时间点(5 days post-anthesis (DPA)、10 DPA、15 DPA和20 DPA)产生的1284份群体转录组数据,记录了渐渗基因在群体中的表达动态特征。作者发现,与稳定表达的渐渗基因相比,容易受到背景基因组影响的渐渗基因更倾向于与纤维品质等表型相关。研究利用开发的多位点模型方法,对纤维发育的4个时间点分别进行了表达数量性状位点(eQTL)的鉴定,并表征了群体中eQTL的分布特点。随后,在渐渗系群体中建立了由渐渗片段介导的遗传调控扰动网络,揭示了纤维发育过程中的动态调控和相互作用(图3)。
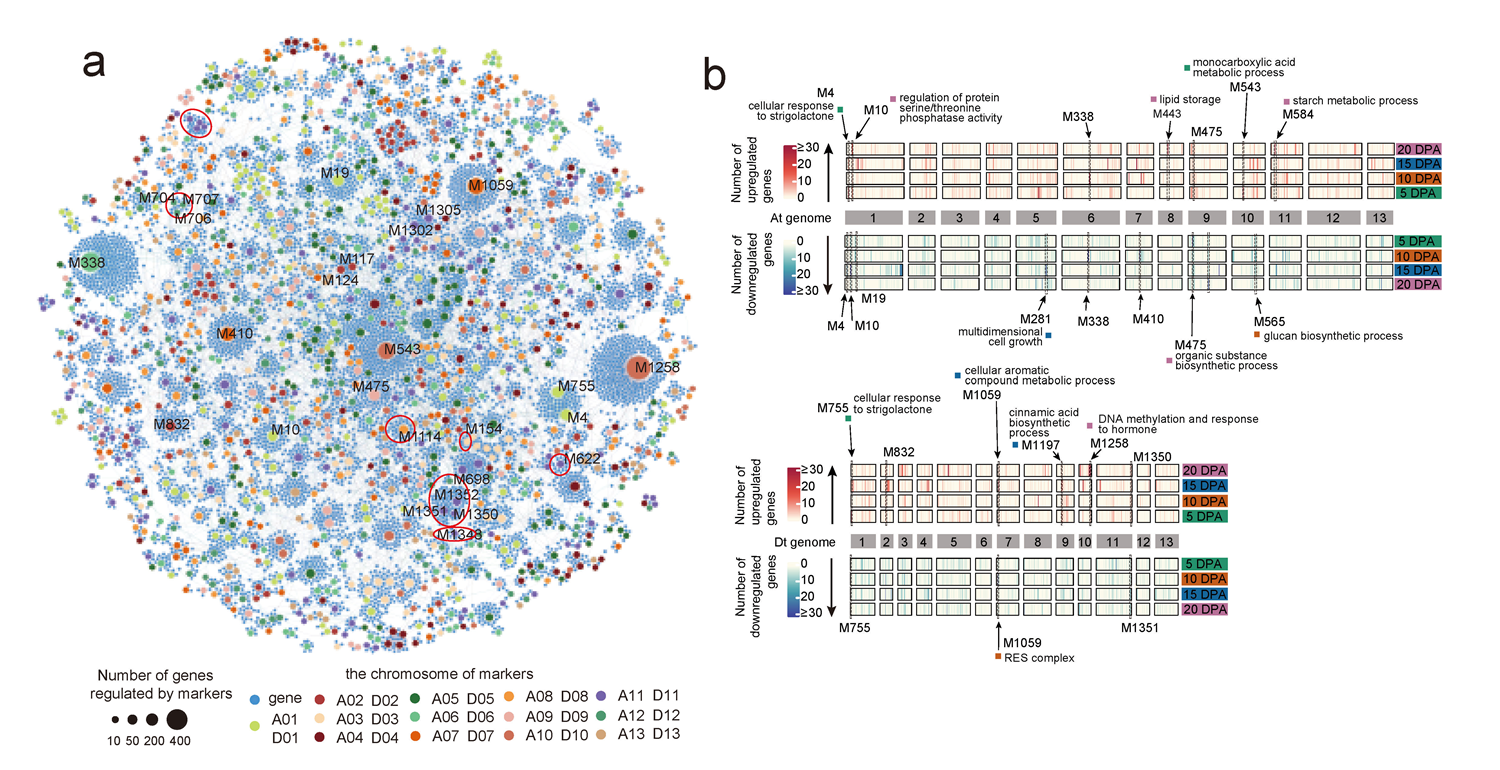
图3. 渐渗介导的遗传调控扰动
最近植物和动物中的遗传分析表明,稀有变异可能导致表型相关的表达失调。为了探讨在棉花中是否存在这一现象,该研究在纤维发育的4个时间点调查了渐渗基因的比例与其表达水平之间的关系。在纤维发育的4个时间点中,渐渗基因的表达呈现极低或极高分布,且倾向于极端低表达。棉花的基因组中包含两个亚基因组At和Dt,这些亚基因组的基因可能由于具有共同的祖先而共享顺式调控序列。因此,探索在同源基因中仅有一个拷贝发生渐渗的情况下,同源基因的表达是否存在差异具有重要意义。在部分的同源基因中,作者观察到渐渗拷贝的极端表达会改变其非渐渗拷贝的基因表达。之后证明这种现象可能与同源基因受到相同的eQTL调控有关,或与一个同源拷贝作为另一个拷贝的调控元件有关(图4)。
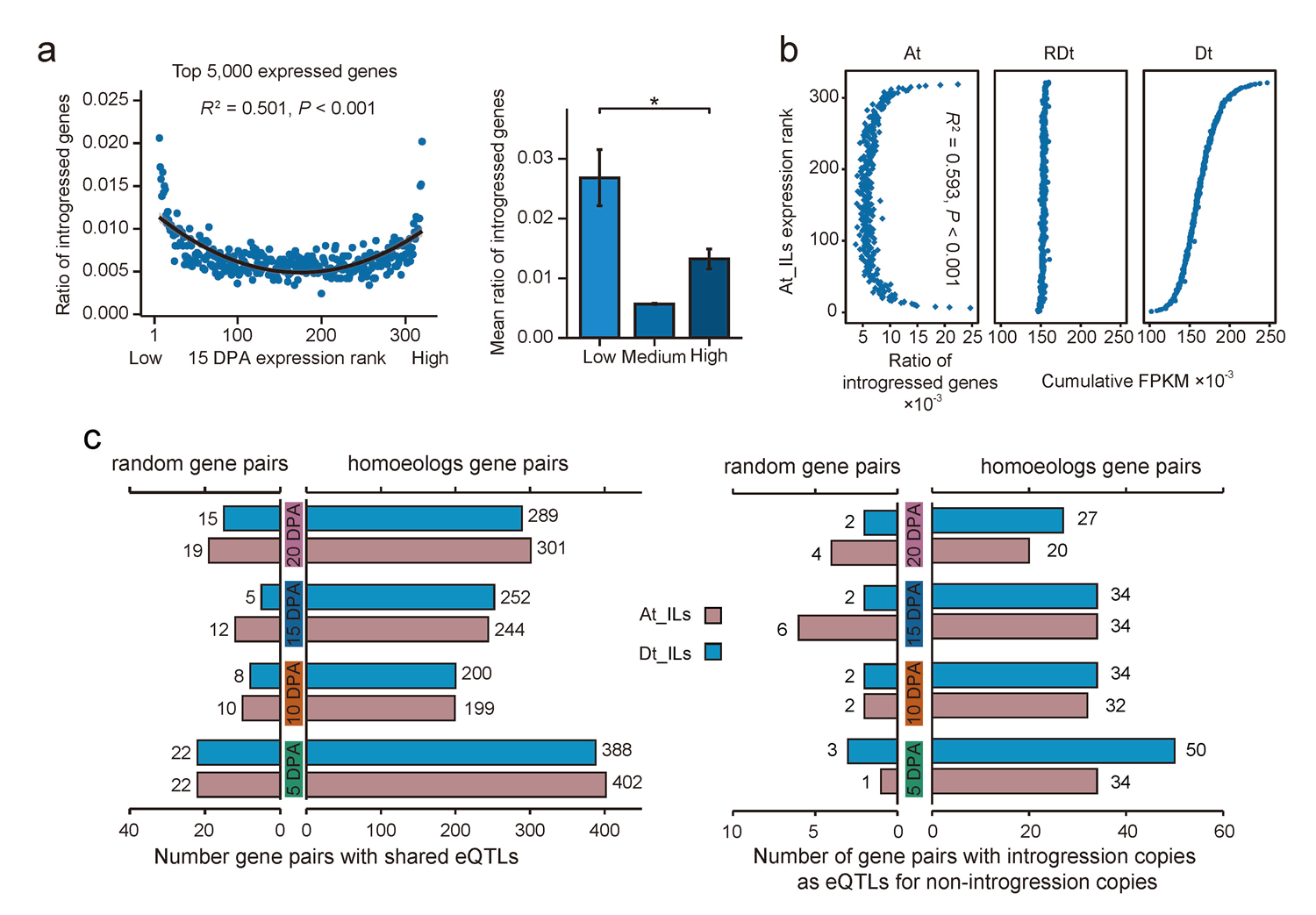
图4. 同源基因的协同表达调控
为了挖掘基因组变异与纤维品质之间的联系,研究鉴定了36个与纤维品质性状相关的QTL。进一步整合eQTL和QTL结果,发现34个QTL中的部分基因能够与不同时期的eQTL共定位。随后,对所有共定位基因在亲本和渐渗系间的表达变化进行表征,构建了群体中渐渗片段的调控分歧模型,揭示了海岛棉基因组片段在陆地棉纤维品质改良中的作用。此外,通过转录组分析,作者挖掘到一个在纤维中表达量存在显著差异的基因GhFLAP1(encodes a fluctuating light acclimation protein,波动光适应蛋白)。随后分析该基因在两个亲本中的序列变异,发现在Emian22的基因区插入了3,639 bp的序列。创制GhFLAP1基因的敲除突变体,并进行纤维品质鉴定,发现与野生型相比,突变体的纤维长度显著降低。
综上所述,该研究提供了大量关于基因组片段和纤维品质之间的遗传调控信息,对于优质棉纤维育种具有重要的参考价值,并有助于加快棉花新品种培育。基因渐渗片段的调控效应分析为研究基因渐渗对其他物种表型变异的贡献提供了重要依据。
华中农业大学作物遗传改良全国重点实验室和湖北洪山实验室的王茂军教授、张献龙院士和美国爱荷华州立大学的Jonathan F. Wendel院士为该论文的共同通讯作者,华中农业大学已毕业博士生陈鑫媛、博士生胡修宝和已毕业博士生李果为该论文的共同第一作者。华中农业大学的章元明教授、林忠旭教授和美国爱荷华州立大学的Corrinne E. Grover等也参与了本项研究。该研究中的田间实验在黄冈市农业科学院棉麦研究所李蔚团队的实验站完成。
【英文摘要】
Interspecific genomic introgression is an important evolutionary process with respect to the generation of novel phenotypic diversity and adaptation. A key question is how gene flow perturbs gene expression networks and regulatory interactions. Here, an introgression population of two species of allopolyploid cotton (Gossypium) to delineate the regulatory perturbations of gene expression regarding fiber development accompanying fiber quality change is utilized. De novo assembly of the recipient parent (G. hirsutum Emian22) genome allowed the identification of genomic variation and introgression segments (ISs) in 323 introgression lines (ILs) from the donor parent (G. barbadense 3–79). It documented gene expression dynamics by sequencing 1,284 transcriptomes of developing fibers and characterized genetic regulatory perturbations mediated by genomic introgression using a multi-locus model. Introgression of individual homoeologous genes exhibiting extreme low or high expression bias can lead to a parallel expression bias in their non-introgressed duplicates, implying a shared yet divergent regulatory fate of duplicated genes following allopolyploidy. Additionally, the IL N182 with improved fiber quality is characterized, and the candidate gene GhFLAP1 related to fiber length is validated. This study outlines a framework for understanding introgression-mediated regulatory perturbations in polyploids, and provides insights for targeted breeding of superior upland cotton fiber.
点击阅读原文
审核人 王茂军