南湖新闻网讯(通讯员 张宇婷)3月28日,华中农业大学油菜团队在Genome Biology发表题为Multi-omics analysis dissects the genetic architecture of seed coat content in Brassica napus的研究论文。该研究在甘蓝型油菜皮壳率的遗传基础解析、新基因挖掘以及种子碳源分配调控网络等方面取得了重要进展。
菜籽油是世界第三大食用植物油,提高油菜籽含油量是油菜育种的重要目标之一。油菜种子主要由种皮和胚组成,胚是储存油脂、蛋白质等营养物质的主要场所,种皮在很多生物过程,如营养运输、控制种子大小以及抵御生物和非生物逆境中都发挥着重要作用。种皮中包含了色素、木质素、纤维等诸多次生代谢物,这些次生代谢物的合成与种皮的正常发育密不可分,其含量决定了种皮的颜色和厚度,并影响皮壳率(种皮重量占种子的比例)。通过调控种皮发育,降低种子皮壳率,可以提高胚的比重,进而提高种子含油量。因此,阐明油菜皮壳率性状形成的遗传基础、挖掘调控种皮发育的关键基因,对提高油菜含油量具有重要意义。
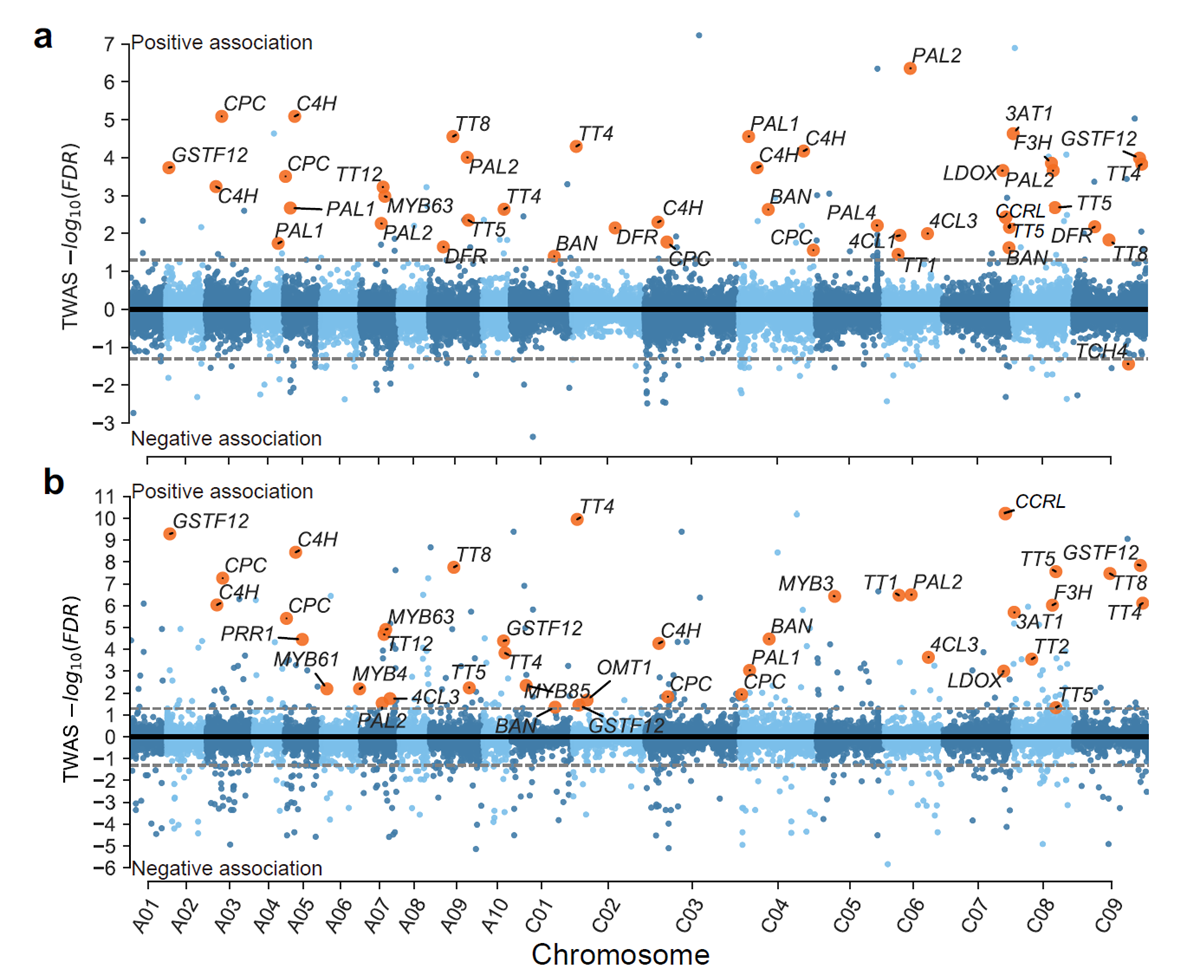
全转录组关联分析鉴定皮壳率显著关联的基因(a 种子发育20天全转录组关联分析;b 种子发育40天全转录组关联分析)
该研究利用382份甘蓝型油菜的基因组重测序数据以及从中选择的257份代表性材料、两个种子发育时期的转录组数据,通过全基因组关联分析(GWAS)和全转录组关联分析(TWAS)等方法对油菜种子皮壳率的遗传基础进行了系统解析。研究通过TWAS分析鉴定到了超过700个基因与皮壳率显著关联,结合基因模块调控网络,发现BnaCCRL和BnaTT8可能通过调控木质素的生物合成从而在种皮发育过程中发挥关键作用。通过GWAS分析,定位了调控皮壳率的3个QTL位点,并且在A09染色体上发现了一个热点QTL qSCC.A09,该QTL可能通过调控BnaTT8影响种皮中木质素的生物合成。
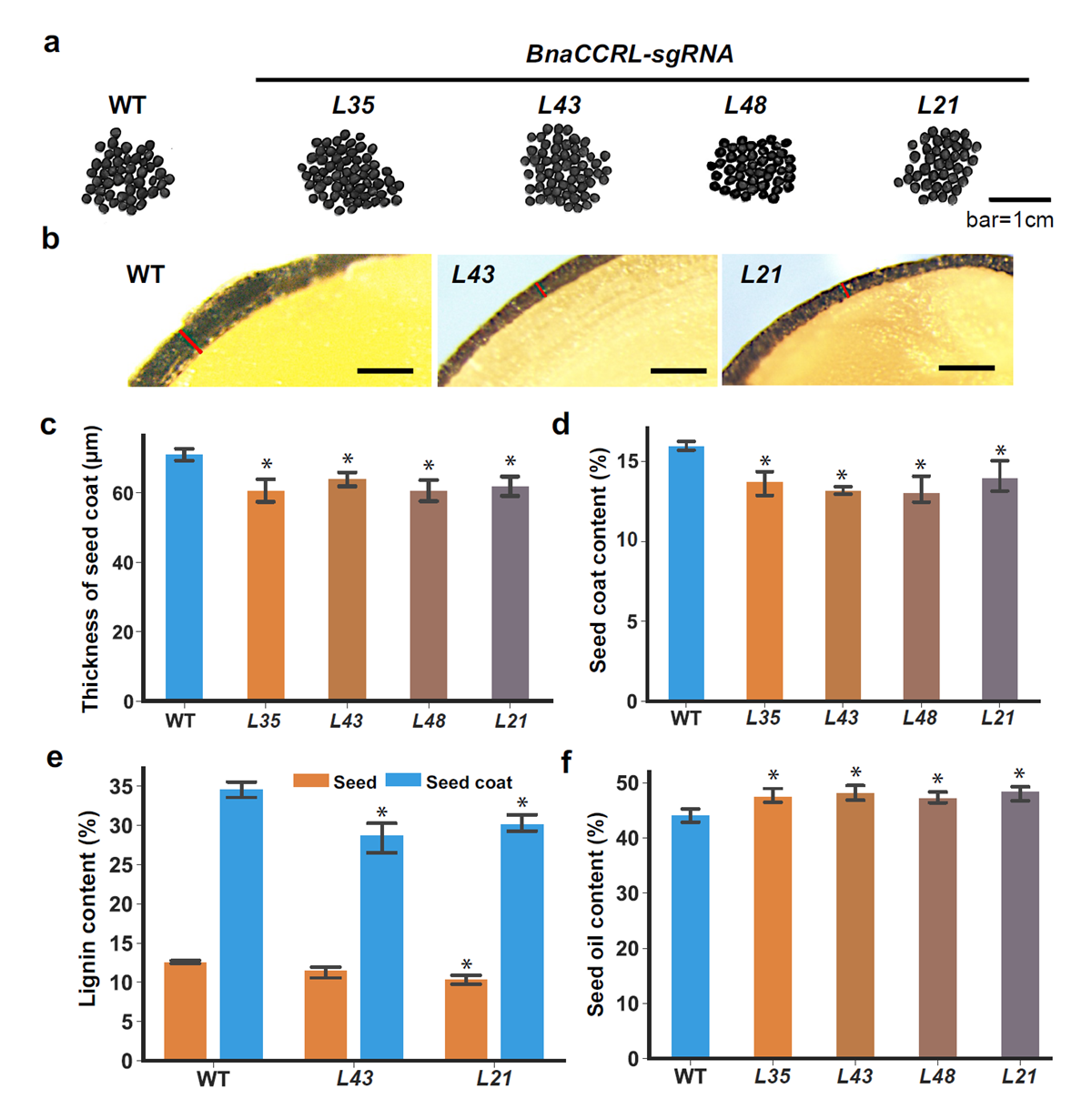
敲除BnaCCRL可降低油菜皮壳率和木质素含量,提高含油量
为了验证BnaCCRL和BnaTT8的基因功能,研究利用基因编辑技术构建了敲除突变体。与野生型相比,BnaCCRL和BnaTT8突变体中木质素含量、皮壳率以及种皮厚度显著降低,而种子含油量显著增加。分析表明,BnaTT8可能通过一般苯丙烷途径间接调控木质素合成,从而影响皮壳率。Cinnamoyl-CoA reductase(CCR,肉桂酰辅酶A还原酶)是参与木质素合成的关键酶,而该研究中鉴定的CCR-like(CCRL)与CCR同源性低于30%,首次通过酶活实验证明了BnaCCRL也具有肉桂酰辅酶A还原酶活性。遗传、生物化学和代谢分析表明,BnaCCRL直接参与种皮木质素生物合成来调控油菜种子的皮壳率。
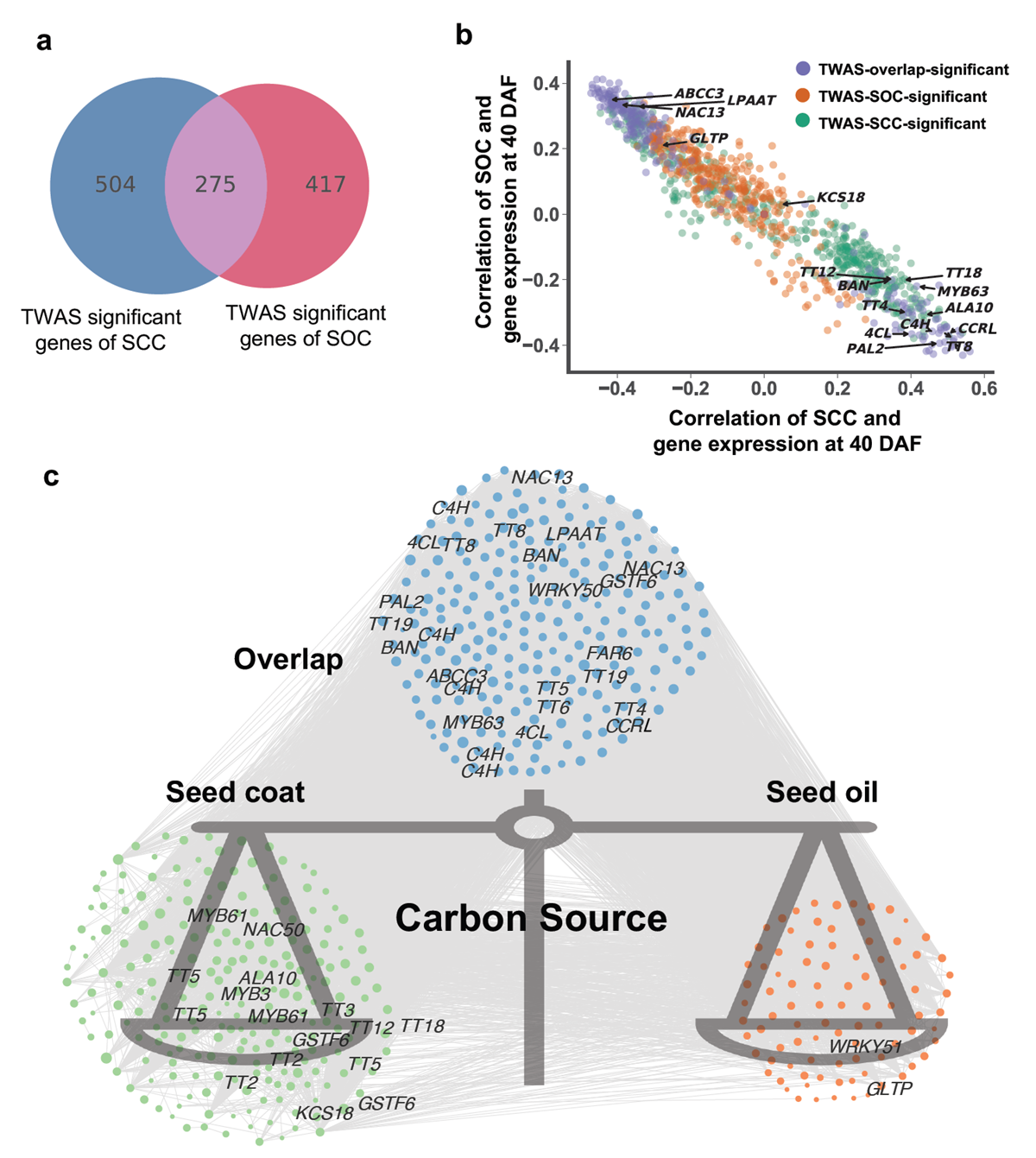
TWAS显著基因在油菜种子的碳源分配存在平衡
甘蓝型油菜种子皮壳率与含油量呈现显著的负相关关系,降低皮壳率有利于种子积累更多的油脂。研究对含油量和皮壳率TWAS显著基因表达进行分析,发现了大量参与苯丙烷途径和油脂合成途径的基因在种子发育过程中影响了油菜种皮和胚之间碳源分配。
该研究全面解析了甘蓝型油菜皮壳率的遗传基础,克隆了两个调控木质素含量和含油量的基因。研究结果对于阐明油菜种皮形成的机制具有重要的理论意义,也为高含油量油菜品种培育提供了基因资源和理论依据。
我校作物遗传改良国家重点实验室博士生张宇婷,硕士生章惠,博士后赵虎为共同第一作者,郭亮教授和姚璇副教授为通讯作者,谢为博教授和胡红红教授也参与了该研究。研究得到了国家自然科学基金(31871658)和湖北洪山实验室重大项目(2021HSZD004)资助。
审核人:姚璇
【英文摘要】
Background
Brassica napus is an important vegetable oil source worldwide. Seed coat content is a complex quantitative trait that negatively correlates with the seed oil content in B. napus.
Results
Here we provide insights into the genetic basis of natural variation of seed coat content by transcriptome-wide association studies (TWAS) and genome-wide association studies (GWAS) using 382 B. napus accessions. By population transcriptomic analysis, we identify more than 700 genes and four gene modules that are significantly associated with seed coat content. We also characterize three reliable quantitative trait loci (QTLs) controlling seed coat content by GWAS. Combining TWAS and correlation networks of seed coat content-related gene modules, we find that BnaC07.CCR-LIKE (CCRL) and BnaTT8s play key roles in the determination of the trait by modulating lignin biosynthesis. By expression GWAS analysis, we identify a regulatory hotspot on chromosome A09, which is involved in controlling seed coat content through BnaC07.CCRL and BnaTT8s. We then predict the downstream genes regulated by BnaTT8s using multi-omics datasets. We further experimentally validate that BnaCCRL and BnaTT8 positively regulate seed coat content and lignin content. BnaCCRL represents a novel identified gene involved in seed coat development. Furthermore, we also predict the key genes regulating carbon allocation between phenylpropane compounds and oil during seed development in B. napus.
Conclusions
This study helps us to better understand the complex machinery of seed coat development and provides a genetic resource for genetic improvement of seed coat content in B. napus breeding.
论文链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02647-5
